Capítulo 15 Normas de reacción e interacción genotipo x ambiente
En capítulos previos hemos desarrollado los principales conceptos de la genética cuantitativa, así como obtenido diferentes resultados cuantitativos a partir del modelo genético básico. Cuando comenzamos a trabajar con dicho modelo, mencionamos la existencia de la interacción entre genotipo y ambiente pero decidimos dejarla fuera de la mayor parte de nuestros desarrollos. El argumento usado es que su importancia es relativamente baja en las condiciones normales asociadas a la producción animal (no así en la vegetal), pero en realidad lo que nos motivó a no considerarla son las complicaciones asociadas a su tratamiento. El presente capítulo vamos a dedicarlo entonces a entender el significado biológico de esta interacción, comprender su significado desde el punto de vista de nuestro modelo genético básico, así como delinear algunas implicancias de esta interacción sobre parámetros genéticos como la varianza aditiva.
Para esto vamos a comenzar por mostrar cuán extendido es el alcance de este efecto y que natural nos resulta su existencia cuando pensamos un poco sobre ello. Vamos a definir conceptos claves como el de plasticidad fenotípica o el de norma de reacción y entender que nuestro concepto de genotipo como algo incambiado y que no reacciona al ambiente es poco natural. Luego pasaremos a analizar formas posibles de estimar su impacto y para eso recurriremos primero al análisis de una característica en dos ambientes. Eso nos remitirá al concepto de respuesta correlacionada, que vimos en el capítulo Correlaciones y Respuesta Correlacionada y la utilidad de la correlación genética como forma de detectar interacciones significativas. Por último, pasaremos a vincular las normas de reacción lineales con los conceptos de genética cuantitativa que vimos en el caso del modelo de un locus con dos alelos y veremos el impacto en la varianza aditiva que puede tener el ambiente.
OBJETIVOS DEL CAPÍTULO
\(\square\) Analizar la interacción genotipo-ambiente, con perspectiva desde el modelo genético básico.
\(\square\) Introducir conceptos como la plasticidad fenotípica y la norma de reacción.
\(\square\) Presentar métodos para estimar el impacto de la interacción genotipo-ambiente.
\(\square\) Analizar ejemplos particulares de interacción genotipo-ambiente, para tomar dimensión de la diversidad de situaciones en las que el fenómeno es de relevancia.
15.1 Plasticidad fenotípica y normas de reacción
Hasta ahora, en lo que hace a la genética cuantitativa, hemos venido trabajando esencialmente con una versión algo simplificada del modelo genético básico. Si bien establecimos desde un principio que el modelo, además de los efectos genéticos y ambientales, podía incorporar los efectos de la interacción entre genotipo y ambiente, hasta ahora logramos evitar razonablemente tener que lidiar con este último factor, que sin duda complica sobremanera muchas de las derivaciones. De hecho, en gran parte de las situaciones que modelamos no parece tan descabellado el ignorar esta interacción. Por ejemplo, cuando el ambiente es relativamente constante o uniforme para todos los individuos, el término de interacción no tendrá significación o importancia práctica. Por ejemplo, si los tambos de una determinada región se encuentran sometidos a las mismas condiciones geográficas, climáticas, etc. y además presentan un manejo muy similar de los animales (algo que parece razonable, ya que los mismos precios de insumos y egresos afectan las decisiones de manejo en forma similar), entonces podemos considerar el “ambiente” como relativamente homogéneo; en estas condiciones, no tiene mucho sentido considerar la interacción entre el genotipo y el ambiente ya que el ambiente es casi el mismo para todos los animales.
Sin embargo, en muchas situaciones los ambientes son claramente contrastantes y ya no es posible evitar la consideración de la interacción entre genotipo y ambiente. Siguiendo con el mismo ejemplo, el uso de la raza Holando (Holstein) se encuentra extendido por todo el orbe, debido a su enorme producción. De hecho, el “tipo” de la raza ha ido cambiando a lo largo del siglo XX, principalmente guiado por los criadores de América del Norte hacia animales más grandes y pesados, adaptados a las condiciones productivas de dichos criadores. Mirada como unidad productiva, cuanto más grande la vaca (dentro de la raza Holando) mayor la expectativa promedio de producción, lo que ha llevado a animales de gran porte (vacas de más de 650 Kg). Estos “genotipos” (en un sentido liberal del término) de animales se encuentran perfectamente adaptados a un ambiente controlado y agradable (en general estabulados buena parte del año), con comida ad libitum y un manejo sanitario excelente, lo que permite que una partición de la energía hacia la producción lechera sea favorecida (es decir, seleccionada favorablemente por los productores).
Por otra parte, cuando estos mismos animales deben producir en otros ambientes, por ejemplo en los trópicos, las condiciones ambientales cambian en forma dramática y las mismas características que eran tan deseables antes ahora parecen claramente perjudiciales. Vacas grandes precisan mucho más alimento que vacas pequeñas y difícilmente puedan mantener una condición corporal razonable ya que además estaban seleccionadas para sacrificar su condición a cambio de la producción de leche. Mucho peor, como mencionamos antes, el calor se genera proporcionalmente al volumen del animal, mientras que se disipa en proporción al área, por lo que animales grandes sufrirán en extremo el calor. Más aún, el manejo sanitario tan cuidadoso que recibieron seguramente evitó el desarrollo de resistencia frente a parásitos, en particular los del nuevo ambiente, que seguramente tiene mayor cantidad y diversidad de los mismos, por lo que apenas podrá mantenerse en condición sanitarias aceptables. Frente a esto, animales de la misma raza, de menor tamaño, criados desde hace varias generaciones en las condiciones de los trópicos seguramente se sentirán más “aclimatados” y adaptados al ambiente exigente, por lo que su producción será razonable, así como su condición corporal y su estado sanitario.
Si por alguna extraña razón llevamos animales Holando adaptados al trópico a producir en América del Norte, seguramente su producción se verá algo incrementada, aunque seguramente ni cerca de la producción de las vacas Holando en uso allí ya que es un animal más pequeño. Ahora, cuando comparamos la producción de los dos “genotipos” (el del trópico y el de América del Norte) en los dos ambientes, claramente la diferencia observada en el trópico será mucho menor que la observada en el ambiente favorable. Dicho de otra forma, desde el punto de vista del modelo no nos alcanza con la suma de los efectos genético y los ambientales para describir el comportamiento de un animal, ya que el mismo dependerá de la combinación específica de genotipo y ambiente; esto es lo que llamamos interacción genotipo-ambiente.
Obviamente que se trata de una situación ejemplo de muchas otras posibles en diferentes tipos de producciones. En general, es más fácil observar el efecto de la interacción entre genotipo y ambiente en plantas que en animales, ya que las primeras tienen muy poca capacidad de escoger individualmente el ambiente. Una animal puede, en cierta medida, elegir área con sombra en un día de calor, o buscar una aguada cuando siente sed, un refugio cuando siente frío, por lo que intenta “controlar” el ambiente externo de tal forma que se encuentre lo más próximo posible a su zona de confort. En cambio, la planta tendrá mucho menos opciones de ir cambiando el ambiente externo a sus necesidades puntuales; si viene seca tendrá que intentar minimizar los daños, pero lo mismo le ocurrirá en la inundación, sol calcinante o ausencia del mismo, invasión de plagas diversas o lo que sea le depare su existencia. Por lo tanto, en general las plantas se encuentran sometidas a condiciones ambientales externas más extremas que los animales y por lo tanto las diferencias en habilidades en cada uno de estos ambientes serán más marcadas.
Existen dos fenómenos distintos que pueden interferir en la simple adición de los efectos genéticos y ambientales: a) la interacción genotipo-ambiente propiamente dicha y que generalmente notamos \(G \times E\) y b) la correlación entre genotipo y ambiente, que notamos como \(r_{GE}\). Como vimos en la sección La Varianza en el modelo genético básico, desde el punto de vista de la varianza fenotípica, el modelo se puede escribir como
\[ \begin{split} V_P=\mathbb{V}(G+E+I_{GxE})=V_G+V_E+V_{GxE}+2\mathrm{Cov}_{GE}= \\ V_A+V_D+V_I+V_E+V_{GxE}+2\mathrm{Cov}_{GE} \end{split} \tag{15.1} \]
Además de los componentes “tradicionales” de la varianza, es decir \(V_A\), \(V_D\), \(V_I\) y \(V_E\), tenemos dos términos adicionales: \(V_{GxE}\) y dos veces \(\mathrm{Cov}_{GE}\). Este último término, \(\mathrm{Cov}_{GE}\), es el que se asocia directamente con la correlación \(r_{GE}\) que mencionamos más arriba. Podemos verlo como un término de “inflación” de la varianza fenotípica observada (cuando es positiva), producto de la co-variación en el mismo sentido entre el genotipo y el ambiente. De los cursos de estadística sabemos que \(\mathrm{Var(x+y)=Var(x)+Var(y)+2\ Cov(x,y)}\), por lo que siempre que las dos variables aleatorias covaríen (es decir, sea distinto de cero el valor de la covarianza), en nuestro caso genotipo y ambiente, la varianza de la suma será diferentes que la suma de las varianzas.
Ejemplos claros de este tipo de efectos es cuando a genotipos más productivos les asignamos ambientes más favorables en forma sistemática, o en el sentido contrario, a los menos productivos les asignamos mejores ambientes. En el primer caso, buscamos explotar al máximo el potencial de cada genotipo, por ejemplo asignando más alimento a las vacas con “mejor genética” (simplemente porque creemos que deben ser más productivas); esto hará que crezca la brecha entre animales y por lo tanto se incremente la varianza fenotípica. Por otro lado, en el segundo caso buscamos una producción más homogénea, por lo que a plantas con peor genética les asignamos más fertilizantes y riego, resultando en mayor homogeneidad y por lo tanto menor varianza fenotípica.
Desde el punto de vista más agronómico, este tipo de covariación o correlación entre genotipo y ambiente puede considerarse como un “ruido” o “contaminación de la información” que muchas veces se puede eliminar haciendo que todos los genotipos se expresen en todos los ambientes. Por ejemplo, cuando comparamos razas de animales (como genotipos diferentes), en general cada raza se encuentra originalmente en determinados ambientes, pero difícilmente todas las razas estén en todos los ambientes de interés. Alcanzaría en principio con tener registros de todas la razas en los ambientes de interés para romper esa asociación específica de genotipos con ambientes.
Por otra parte, el segundo efecto mencionado, la varianza de la interacción entre genotipo y ambiente es de una base mucho más biológica, ya que lo es la interacción misma y generalmente imposible de eliminar. Cuando pensamos en una característica de interés productivo, por ejemplo la producción de leche, tendemos a pensar que los genes que la gobiernan son aquellos que tienen que ver con el funcionamiento de la glándula mamaria, cosa que en principio suena razonable. Por lo tanto, estos genes se expresarán no importa en qué ambiente, mientras que la vaca se encuentre en lactancia. Sin embargo, la vaca es la entidad biológica relevante para la producción de leche, no la ubre; la vaca sometida a estrés térmico tendrá disparados un conjunto de mecanismos para la homeostasis que perturbarán enormemente la capacidad de la ubre de utilizar los recursos del animal.
En gran parte de lo que hemos trabajado en la parte de genética cuantitativa nos hemos mantenido con la idea de que el genotipo y el ambiente no presentan interacción, al menos significativa. Es decir, un genotipo siempre aporta lo mismo al fenotipo de una característica. Sin embargo, como acabamos de ver, esto no siempre es así. De hecho, un genotipo puede reaccionar en forma diferentes a diferentes ambientes o en forma más general, a un gradiente ambiental (una variación continua del ambiente, como puede ser la humedad del suelo, el nivel de luz o de radiación solar, etc.). En general un fenotipo es la respuesta visible a través de una característica particular (producción de algo, largo de otra cosa, color de otra diferente, etc.) de la expresión de un conjunto de proteínas (estructurales, enzimáticas, etc.) todas reguladas por un conjunto de genes, que a su vez reciben información del medio externo a través de un conjunto de receptores y de vías de transmisión de la información. Por lo tanto, no debería sorprendernos para nada que un determinado “genotipo” responda en forma diferente, para la misma característica, a diferentes condiciones ambientales. Precisamente, la plasticidad fenotípica es la capacidad de un organismo de producir fenotipos diferentes en respuesta a cambios en el ambiente.
Se llama plasticidad fenotípica a la capacidad de realizar cambios en el comportamiento, la morfología y la fisiología de un organismo en respuesta a cambios en el ambiente. La plasticidad fenotípica abarca todos los tipos de cambios inducidos por el medio ambiente (por ejemplo, morfológicos, fisiológicos, de comportamiento, fenológicos) y que pueden o no ser permanentes a lo largo de la vida de un individuo.
Usualmente es más fácil de observar plasticidad fenotípica en las plantas como mecanismo de adaptación a la variabilidad ambiental a las que se ven sometidas. Ejemplos claros incluyen la asignación de más recursos a las raíces en suelos que contienen bajas concentraciones de nutrientes, el tiempo de floración, así como el área y espesor de las hojas. En algunas plantas, por ejemplo del género Convolvulus González and Gianoli (2004), mientras que las hojas que crecen en ambiente sombreado muestran una gran superficie y son de poco espesor para maximizar la captura de la luz, en ambientes soleados muestran hojas de menor superficie y más gruesas. También el diente de león (del género Taraxacum) es bien conocido por mostrar una considerable plasticidad en su forma cuando crece en ambientes soleados frente a los sombreados.
El concepto de plasticidad fenotípica se encuentra estrechamente relacionado al concepto de norma de reacción, que sería la expresión de este concepto para una característica fenotípica determinada, en un genotipo y en gradiente ambiental particular.
Se conoce como norma de reacción a la función que relaciona la expresión fenotípica media de un genotipo en diferentes ambientes. Para cada característica fenotípica, genotipo y variable ambiental puede existir una norma de reacción específica.
La norma de reacción de un genotipo dado es su rango de respuestas fenotípicas a lo largo de un gradiente ambiental. El concepto fue introducido por Richard Woltereck en 1909 (Woltereck (1909)) en un trabajo con Dafnias (un género de crustáceos planctónicos del orden Cladocera) y se ilustra en la Figura 15.1. En un gradiente ambiental, marcado por el eje de las abscisas, los dos genotipos A y B reaccionan de manera opuesta en la expresión fenotípica de una característica. Por ejemplo, la característica fenotípica podría ser el rendimiento y el gradiente ambiental podría ser la humedad del suelo. Mientras que para el genotipo A el rendimiento aumenta en forma aproximadamente lineal con la humedad, para el genotipo B el rendimiento decrece de la misma forma con la humedad.
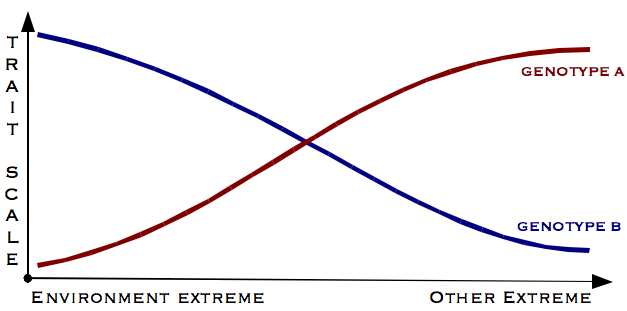
Figura 15.1: Ilustración del concepto de norma de reacción. En un gradiente ambiental representado en las abscisas ambos genotipos reacción de forma opuesta (y aproximadamente lineal) ya que la respuesta fenotípica varía en sentido opuesto (Dominio Público, https://en.wikipedia.org/w/index.php?curid=3925138).
Los conceptos de plasticidad fenotípica y de norma de reacción están estrechamente vinculados al concepto de canalización, un término acuñado en 1942 por Conrad Hal Waddington (Waddington (1942)) con la idea de conceptualizar la robustez que demuestran ciertos procesos y reacciones involucradas en el desarrollo de los organismos y que pese a ser apartadas (ligeramente) de las condiciones normales igual llevan al mismo resultado.
Se llama canalización a una medida de la capacidad de una población para producir el mismo fenotipo independientemente de la variabilidad de su ambiente o genotipo. Es una forma de robustez evolutiva.
De alguna manera, la canalización juega un papel antagónico con la plasticidad fenotípica, amortiguando cambios “ambientales” para que el fenotipo continúe en una región óptima. Waddignton prefirió la utilización de este nuevo término para distinguir la robustez biológica (más ligada al concepto actual de resiliencia) del concepto tradicional en ingeniería. El término proviene de una analogía donde se puede pensar el paisaje epigenético del desarrollo como un valle rodeado por elevaciones y en el que las características canalizadas seguirán el curso a través del valle hacia su “destino” (el fenotipo objetivo). El concepto puede ilustrarse como lo muestra la Figura 15.2. Mientras que el genotipo A muestra cambios importantes en el fenotipo (ordenadas) de acuerdo al gradiente ambiental (en las abscisas), el genotipo B muestra una distribución bimodal fenotípica, de acuerdo a la región del gradiente ambiental. En cada una de estas regiones, sin embargo, los cambios fenotípicos son mínimos a pesar del cambio amplio en el ambiente.
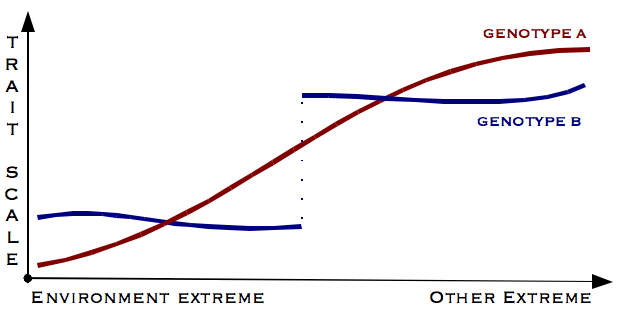
Figura 15.2: Norma de reacción para ilustrar el concepto de canalización. Mientras que el genotipo A muestra una amplia respuesta fenotípica al gradiente ambiental, el genotipo B muestra dos regiones disjuntas del gradiente en las que, en ambas, el fenotipo no cambia mayormente pese a los cambios ambientales; es decir, el genotipo B se encuentra canalizado. (Dominio Público, https://commons.wikimedia.org/w/index.php?curid=1223392).
Desde el punto de vista molecular la canalización podría estar actuando como un capacitor evolutivo, manteniendo diversos cambios a nivel genómico sin expresión en el fenotipo, hasta que por algún motivo el mismo se descarga (la analogía es con el funcionamiento de un capacitor en los circuitos eléctricos, donde el mismo es capaz de almacenar energía mientras se carga). Un ejemplo de un gen con esta función podría ser la proteína HSP90 (“Heat Shock Protein 90”, las HSP son un conjunto de proteínas que originalmente se asociaron a la expresión como respuesta al estrés), que cumple una función de “chaperona” (ayudan al plegado de otras proteínas). Se ha demostrado en diversas especies que la mutación de esta proteína está asociada al incremento en la variabilidad observada en diferentes características fenotípicas, que estarían canalizadas por la forma normal de HSP90.
PARA RECORDAR
Existen dos fenómenos distintos que pueden interferir en la simple adición de los efectos genéticos y ambientales: a) la interacción genotipo-ambiente propiamente dicha y que generalmente notamos \(G\)x\(E\) y b) la correlación entre genotipo y ambiente, que notamos como \(r_{GE}\).
En términos de varianzas, podemos escribir ahora el modelo genético básico como: \(V_P=V_A+V_D+V_I+V_E+V_{GxE}+2\mathrm{Cov}_{GE}\) dónde el término \({Cov}_{GE}\) se asocia directamente con la correlación \(r_{GE}\). Podemos verlo como un término de “inflación” de la varianza fenotípica observada (cuando es positiva), producto de la co-variación en el mismo sentido entre el genotipo y el ambiente.
Llamaremos plasticidad fenotípica a la capacidad de realizar cambios en el comportamiento, la morfología y la fisiología de un organismo en respuesta a cambios en el ambiente. La plasticidad fenotípica abarca todos los tipos de cambios inducidos por el medio ambiente y que pueden o no ser permanentes a lo largo de la vida de un individuo.
Se conoce como norma de reacción a la función que relaciona la expresión fenotípica media de un genotipo en diferentes ambientes. Para cada característica fenotípica, genotipo y variable ambiental puede existir una norma de reacción específica.
Llamaremos canalización a una medida de la capacidad de una población para producir el mismo fenotipo independientemente de la variabilidad de su ambiente o genotipo. Es una forma de robustez evolutiva. Desde el punto de vista molecular la canalización podría estar actuando como un capacitor evolutivo, manteniendo diversos cambios a nivel genómico sin expresión en el fenotipo, hasta que por algún motivo el mismo se descarga y el cambio es apreciable.
15.2 Interacción GxE en dos ambientes
Cuando en lugar de considerar un gradiente ambiental, es decir una variación continua del ambiente, consideramos una serie discreta de ambientes contrastantes, la situación puede simplificarse bastante, particularmente en el caso de dos ambientes. En muchos casos, si bien existe un gradiente ambiental subyacente, nos interesa contrastar dos ambientes en particular, por ejemplo, la expresión de una característica en clima templado versus su expresión en los trópicos. Más específico aún, podemos analizar el uso de una raza cebuina en nuestro país, comparando los resultados de varias razas británicas y de la raza cebuina en nuestro país y en la región de origen de la raza cebuina. Otro ejemplo podría ser analizar la producción de diferentes variedades de maíz en el “Medio Oeste Estadounidense” y en nuestro país.
Un rango de comportamientos posibles de estos experimentos puede apreciarse en la Figura 15.3, donde la expresión fenotípica de tres “genotipos” diferentes, identificados por colores diferentes, se muestran en dos ambientes diferentes, uno pobre y el otro favorable.
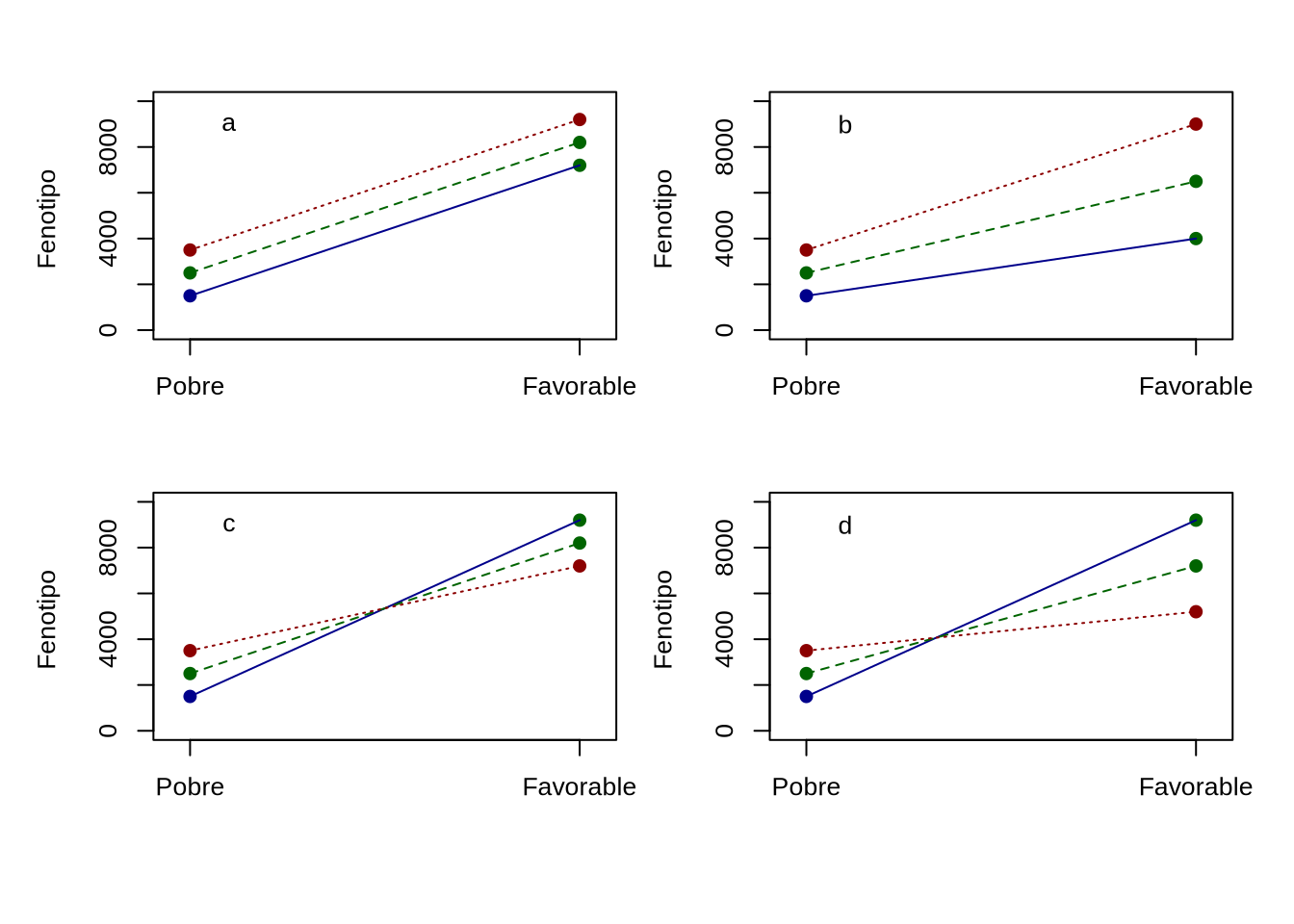
Figura 15.3: Representación esquemática de la interacción genotipo ambiente. Tres genotipos expresan una característica fenotípica cuantitativa (ordenadas) en dos ambientes diferentes (abscisas), un ambiente pobre y uno favorable. (a) Existe un desplazamiento de la característica entre los dos ambientes que es constante para todos los genotipos (alcanza con sumar la diferencia entre ambientes), por lo que no existe interacción entre genotipo y ambiente. (b) La interacción entre genotipo y ambiente se debe a un cambio de escala. (c) La interacción genotipo-ambiente está asociada a un cambio en el ordenamiento de los genotipos (y a un desplazamiento constante de los ambientes). (d) La interacción genotipo-ambiente está asociada a un re-ordenamiento y a un cambio de escala.
En la situación representada en (a), los fenotipos correspondientes a los tres genotipos se desplazan la misma cantidad en el ambiente favorable respecto al ambiente pobre, por lo que genotipo y ambiente son aditivos entre ellos (su suma es el fenotipo) y por lo tanto no existe interacción entre genotipo y ambiente. En la situación mostrada en (b), se mantiene el ordenamiento relativo entre los genotipos (el rojo superior al verde y este superior al azul) en los dos ambientes, pero las distancias entre ellos no son idénticas en los dos ambientes, sino que resultan de multiplicar la distancia original for un factor. Por lo tanto, en este caso estamos tratando con un cambio de escala entre los dos ambientes. Por otra parte, la situación representada en (c) implica un cambio en el ordenamiento de los genotipos (las líneas se cruzan), aunque no hay un cambio de escala ya que la varianza fenotípica es igual en ambos ambientes. Por último, la situación representada en (d) implica tanto un re-ordenamiento de los genotipos como un cambio de escala ya que existe mayor variabilidad fenotípica en el ambiente favorable.
Una simplificación notable en el tratamiento de la interacción entre genotipo y ambiente proviene de la idea de Douglas S. Falconer (1952) de considerar la expresión de la característica en dos ambientes como si se tratase de dos características diferentes y que presentan, por lo tanto, una correlación genética (aditiva). La idea nos remite directamente al “path analysis” de Sewall Wright y a la sección La relación entre las correlaciones fenotípica, genética y ambiental. La idea se ilustra en la Figura 15.4, donde la misma característica se presenta en dos tratamientos diferentes, \(T1\) y \(T2\).
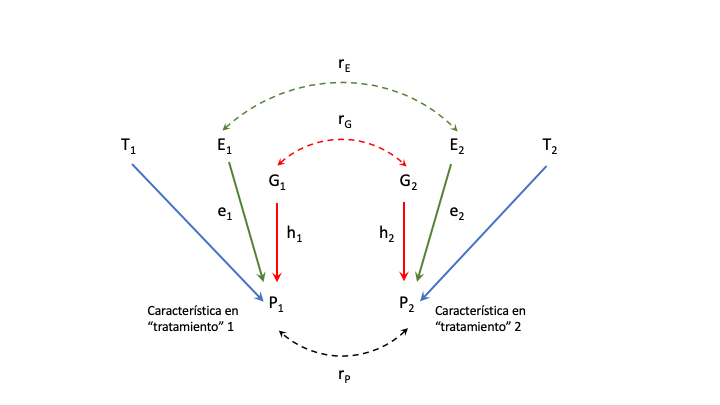
Figura 15.4: Diagrama para el estudio de la interacción genotipo-ambiente en dos ambientes, considerando la misma como una correlación genética entre dos características diferentes. En este diagrama \(T_1\) y \(T_2\) son los dos tratamientos diferentes, que pueden ser considerados como “macroambientes”., mientras que \(E_1\) y \(E_2\) son ambientes con una correlación entre ellos establecida en el modelo.
Podemos pensar en estos tratamientos como dos condiciones nutricionales diferentes, o de exposición diferente a desafíos sanitarios, o lo que se nos ocurra como los dos ambientes contrastantes donde queremos explorar el tema de interacción genotipo ambiente.
Si observamos la figura, hay tres fuentes de variación que afectan a cada uno de los dos fenotipos: los dos genotipos, \(G_1\) y \(G_2\); las diferencias ambientales dentro de cada tratamiento, \(T_1\) y \(T_2\); y las diferencias ambientales no asociadas a los tratamientos, \(E_1\) y \(E_2\). Los dos fenotipos, que en realidad es la misma característica medida en dos ambientes diferentes, están correlacionados por una vía genética y, posiblemente, por una vía ambiental. Por otra parte, los dos genotipos, \(G_1\) y \(G_2\), están conectados por una correlación, \(r_G\).
Como vimos en el capítulo Correlaciones y Respuesta Correlacionada, esta correlación genética, junto con los coeficientes de paso, \(h_1\) y \(h_2\) (las raíces cuadradas de las dos heredabilidades), que relacionan el genotipo con el fenotipo son todo lo que se necesita para la solución del problema de determinar el coeficiente de correlación genética, ya que de acuerdo a la ecuación (12.32), si las intensidades de selección son las mismas en las dos características, entonces
\[ \begin{split} \mathrm{\frac{RC_2}{R_2}=\frac{h_1}{h_2}\ r_{A_{12}}} \end{split} \]
y un estimador razonable del valor absoluto del coeficiente de correlación genética era el dado por la ecuación (12.33)
\[ \begin{split} \mathrm{r^2_{A_{12}}=\frac{RC_1}{R_1}\frac{RC_2}{R_2}}\ \therefore\ \mathrm{\|r_{A_{12}}\|=\sqrt{\frac{RC_1}{R_1}\frac{RC_2}{R_2}}} \end{split} \tag{12.33} \]
Al tratarse como dos características diferentes, podemos considerar que los ambientes actúan de diferente forma en cada una y lo mismo la genética. Si desde el punto de vista genético el ordenamiento es exactamente igual en los dos ambientes y las distancias proporcionales, entonces la correlación genética debería ser igual a uno. Por el contrario, si de alguna manera el orden de los fenotipos se altera o aún las distancias entre ellos dejan de ser proporcionales, entonces la correlación será inferior a uno.
Una de las dificultades mayores con esta idea es la de determinar el fenotipo en dos tratamientos diferentes al mismo tiempo o al menos en tiempos suficientemente similares como que no sean importantes los factores de confusión. Por ejemplo, es imposible por este método determinar la existencia de interacción genotipo-ambiente en peso a los 18 meses para animales criados en confinamiento vs criados a campo, ya que un animal no puede estar en los dos estados (tratamientos) al mismo tiempo (excepto para el gato de Schrödinger, pero en este caso es bastante difícil medir los fenotipos). Para otras características, que de alguna manera se repiten en el animal, por ejemplo las lactancias, podemos ensayar los animales en las dos condiciones, pero resulta un factor importante de confusión la historia previa del animal antes de cada lactancia y además existe el factor de confusión que es el número de lactancia (cuando nos referimos a producción del animal).
Una alternativa razonable es la experimentación con grupos de individuos con algún tipo de vinculación de parentesco entre ellos, como la unidad básica, en lugar del individuo. Es decir, podemos dividir a la mitad cada grupo (familia) y dejar una mitad en un tratamiento y la otra mitad en el otro tratamiento. Por comodidad, a veces vamos a llamar macroambiente al tratamiento, ya que se trata de un “ambiente” que afecta a todo un grupo de individuos (el que está sometido a ese tratamiento, que obviamente no es genético). En lo que sigue, en esta y en la próxima sección, vamos a seguir muy estrechamente el tratamiento del tema que le dan Michael Lynch and Walsh (1998), por lo que nuevamente apelaremos a su notación.
Supongamos un experimento en el que pretendemos evaluar la interacción genotipo-ambiente entre dos macroambientes. Para eso, vamos a dividir todos los grupos genotípicos de interés (que pueden estar representados, por ejemplo, por familias) en dos, una mitad expresando la característica en un macroambiente, la otra mitad en el otro. En el presente contexto, podemos describir el fenotipo de un individuo que participa en nuestro experimento como
\[ \begin{split} z_{ijk}=\mu+G_j+I_{ij}+E_{g,i}+E_{s,ijk} \end{split} \tag{15.2} \]
donde \(\mu\) es la media general, promediada sobre todos los genotipos en todos los ambientes, \(G_j\) es el efecto del \(j\)esimo grupo genotípico, promediado sobre todos los ambientes, \(E_{g,i}\) es el efecto promedio del \(i\)esimo ambiente sobre todos los genotipos, \(I = {ij}\) es el efecto de interacción entre el genotipo \(j\) y el ambiente \(i\) y \(E_{s,ijk}\) son los efectos ambientales particulares (error) de la réplica \(k\) del genotipo \(j\) en el ambiente \(i\). La notación de la letra \(g\) en \(E_{g,i}\) se refiere a que se trata de un efecto ambiental general (macroambiente, tratamiento \(T\) en Douglas S. Falconer (1952)), mientras que la letra \(s\) en \(E_{s,ijk}\) se refiere al ambiente específico (\(E\) en Douglas S. Falconer (1952)).
De acuerdo con este modelo, el efecto del macroambiente \(i\) podemos escribirlo como el desvío de la media de los todos los grupos en dicho macroambiente respecto a la media general, es decir
\[ \begin{split} E_{g,i}=\mu_i-\mu \end{split} \tag{15.3} \]
Del mismo modo, de acuerdo a la definición dada previamente sobre el modelo, el efecto particular de un grupo genético estará dada por el desvío de la media de ese grupo en los dos macroambientes respecto a la media general, es decir
\[ \begin{split} G_j=\mu_j-\mu \end{split} \tag{15.4} \]
Por otra parte, el desvío correspondiente a la interacción entre el genotipo \(j\) y el ambiente \(i\) es igual a la media de la combinación de dichos genotipo en ese ambiente, respecto a la media general, a la media del genotipo y a la media del ambiente, es decir
\[ \begin{split} I_{ij}=\mu_{ij}-\mu-E_{g,i}-G_j=\mu_{ij}-\mu-[\mu_i-\mu]-[\mu_j-\mu]=\\ =\mu_{ij}+\mu-\mu_i-\mu_j \end{split} \tag{15.4} \]
Cuando no existe interacción entre genotipo y ambiente \(I_{ij}=0\), por lo que
\[I_{ij}=\mu_{ij}+\mu-\mu_i-\mu_j=0 \Leftrightarrow \mu_{ij}=-\mu+\mu_i+\mu_j \Leftrightarrow\] \[\mu_{ij}=-\mu+\mu_i+\mu_j+\mu-\mu=\mu+(\mu_i-\mu)+(\mu_j-\mu) \Leftrightarrow\]
\[ \begin{split} \mu_{ij}=\mu+G_j+E_{g,i} \end{split} \tag{15.5} \]
o lo que es lo mismo, la media del genotipo \(j\) en el ambiente \(i\) será igual a la suma de la media general, el efecto general del genotipo \(j\) y el efecto general del ambiente \(i\). Desde el punto de vista gráfico esto significa que, con dos macroambientes, las líneas que unen a los genotipos en los dos ambientes serán paralelas.
Ejemplo 15.1
En un experimento se determinó que la producción de dos variedades determinadas de maíz en (kg/ha) son funciones lineales de la cantidad de agua disponible en el suelo en un momento específico del crecimiento, al menos en un rango muy estrecho, entre \(18\%-22\%\). Las funciones que describen las producciones de las dos variedades (A y B) en función de la humedad del suelo en el momento estudiado son:
A: \(P_A=[7800+h\% \times 250] \text{ kg}\)
B: \(P_B=[5300+h\% \times 375] \text{ kg}\)
Graficar la producción de los dos cultivos en el gradiente de humedad establecido. ¿Existe interacción genotipo-ambiente? Determinar el punto en que ambas producciones son iguales. ¿Qué variedad nos conviene más si suponemos un ambiente relativamente seco en esa etapa del cultivo?
Se trata de dos funciones lineales, por lo que alcanza con determinar un par de puntos para cada función y luego unirlos. Por ejemplo, en forma completamente arbitraria podemos elegir los puntos de \(19\%\) y \(21\%\), lo que nos deja con los siguiente pares de puntos para A y B:
| \(\text{Variedad}\) | \(\text{Humedad}\) | \(\text{Rendimiento (kg/ha)}\) |
|---|---|---|
| \(\textbf{A}\) | \(19\%\) | \([7800+19 \times 250] \text{ kg}=12.550 \text{ kg}\) |
| \(\textbf{A}\) | \(21\%\) | \([7800+21 \times 250] \text{ kg}=13.050 \text{ kg}\) |
| \(\textbf{B}\) | \(19\%\) | \([5300+19 \times 375] \text{ kg}=12.425 \text{ kg}\) |
| \(\textbf{B}\) | \(21\%\) | \([5300+21 \times 375] \text{ kg}=13.175 \text{ kg}\) |
La gráfica resultante se puede apreciar en la Figura 15.5. Claramente, las dos rectas no son paralelas, para lo que alcanzaba observar que los dos coeficientes de regresión son diferentes (\(b_A=250\), \(b_B=375\), ambas en \(\text{ kg}\)/%), por lo que se observa interacción genotipo-ambiente en el rango considerado. Pero además, las rectas se cruzan, por lo que a ambos lados del punto de corte el ordenamiento por rendimiento cambia: mientras que a la izquierda del punto de corte la variedad A es más productiva, a la derecha del mismo la variedad B es la de mayor rendimiento.
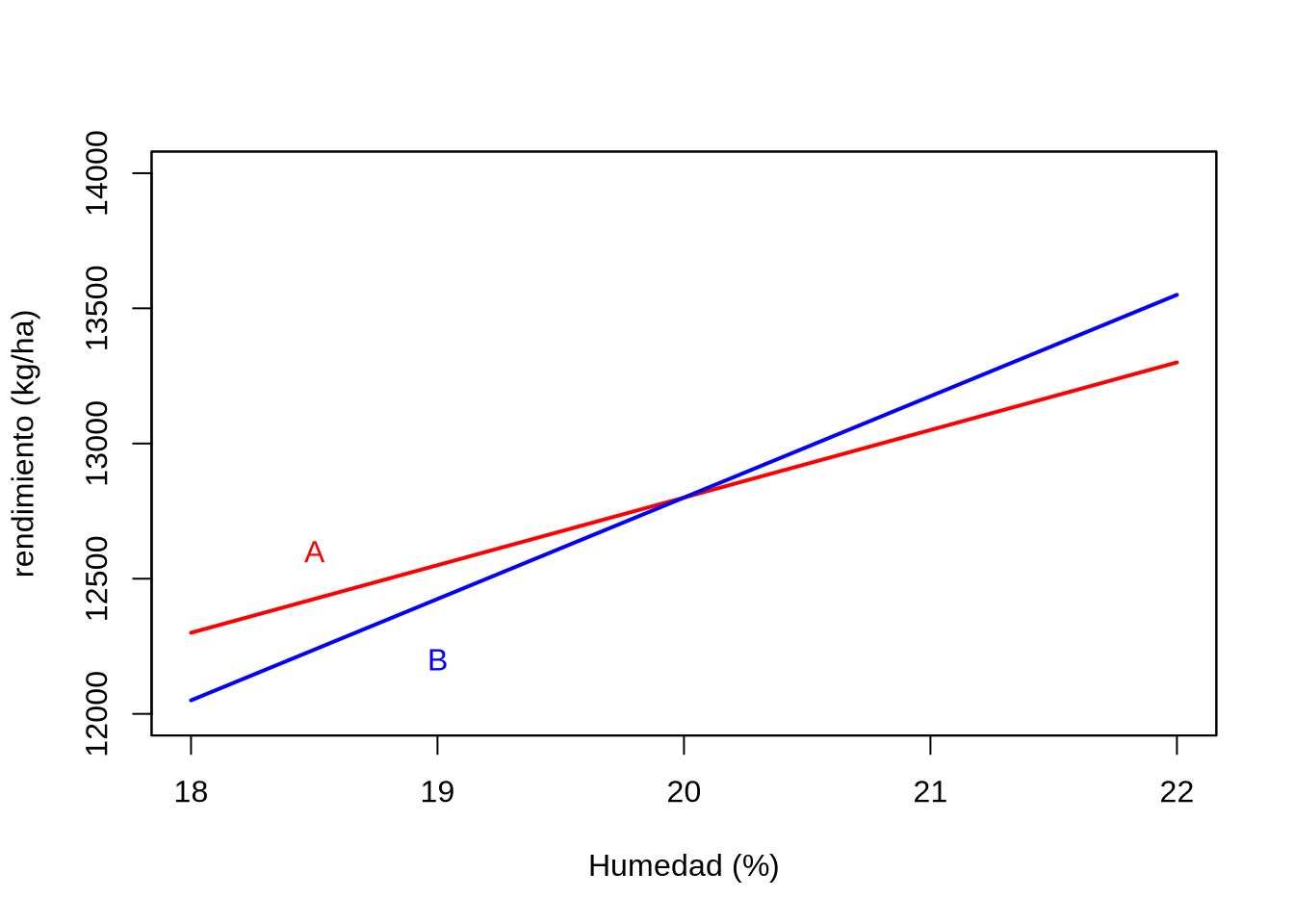
Figura 15.5: Rendimiento en función de la humedad del suelo para dos variedades de maíz, la variedad A en rojo y la variedad B en azul. Se aprecia claramente que las dos líneas no son paralelas (lo que alcanza para determinar la existencia de interacción genotipo-ambiente), peroa además las mismas se cortan en el intervalo considerado, lo que indica un cambio de “ranking” en el mismo.
El punto de corte es fácil de calcular: alcanza igualar ambas rectas y determinar a que porcentaje de humedad corresponde la solución, es decir
\[P_A=[7800+h\% \times 250] \text{ kg}=[5300+h\% \times 375] \text{ kg}=P_B \Leftrightarrow\] \[(7800-5300)=h\% (375-250) \Leftrightarrow h\% = \frac{7800-5300}{375-250}=\frac{2500}{125}=20\]
Para verificar alcanza con sustituir \(h\%=20\%\) en las ecauciones de rendimiento, lo que nos deja con
\[P_A=[7800+h\% \times 250] \text{ kg}=12.800 \text{ kg}\] \[P_B=[5300+h\% \times 375] \text{ kg}=12.800 \text{ kg}\]
La variedad más conveniente en un ambiente relativamente seco parece ser la variedad A, ya que es la que muestra mayor rendimiento por debajo de \(20\%\) de humedad en el suelo.
PARA RECORDAR
Si bien existe un gradiente ambiental subyacente, usualmente nos interesa contrastar dos (o más) ambientes en particular, considerándolos como una serie discreta.
Una simplificación plausible en el tratamiento de la interacción entre genotipo y ambiente proviene de la idea de considerar la expresión de la característica en dos ambientes como si se tratase de dos características diferentes y que presentan, por lo tanto, una correlación genética (aditiva).
Un estimador razonable del valor absoluto del coeficiente de correlación genética será: \(\mathrm{\|r_{A_{12}}\|=\sqrt{\frac{RC_1}{R_1}\frac{RC_2}{R_2}}}\). Al tratarse como dos características diferentes, se puede considerar que los ambientes actúan de diferente forma en cada una y lo mismo la genética. Si desde el punto de vista genético el ordenamiento es exactamente igual en los dos ambientes y las distancias proporcionales, entonces la correlación genética debería ser igual a uno.
En ocasiones donde el uso de un solo individuo podría dar lugar a errores en el resultado, parece razonable la experimentación con grupos de individuos con algún tipo de vinculación de parentesco entre ellos como la unidad básica, en lugar del individuo.
Si pretendiéramos evaluar la interacción genotipo-ambiente entre dos macroambientes y para ello dividiéramos todos los grupos genotípicos de interés, podríamos describir el fenotipo de un individuo que participa en nuestro experimento como: \(z_{ijk}=\mu+G_j+I_{ij}+E_{g,i}+E_{s,ijk}\)
De acuerdo con ese modelo, el efecto del macroambiente \(i\) podemos escribirlo como el desvío de la media de los todos los grupos en dicho macroambiente respecto a la media general, es decir: \(E_{g,i}=\mu_i-\mu\) y consecuentemente, el efecto particular de un grupo genético estará dada por el desvío de la media de ese grupo en los dos macroambientes respecto a la media general tal que: \(G_j=\mu_j-\mu\).
Además, el desvío correspondiente a la interacción entre el genotipo \(j\) y el ambiente \(i\) es igual a la media de la combinación de dichos genotipo en ese ambiente, respecto a la media general, a la media del genotipo y a la media del ambiente, es decir: \(I_{ij}=\mu_{ij}+\mu-\mu_i-\mu_j\)
15.3 Correlación genética a través de dos ambientes
Para simplificar la notación y teniendo en cuenta que en el “error” pueden participar alguna otra fuente genética cuando el fenotipo es de un grupo de individuos, vamos a usar la notación \(e_{ijk}\) en lugar de \(E_{s,ijk}\). Con esto en mente, los fenotipos de dos individuos \(k\) y \(l\), ambos pertenecientes al mismo grupo genotípico \(j\), para la característica de interés en los dos ambientes, \(1\) y \(2\) podemos escribirlos como
\[ \begin{split} z_{1jk}=\mu+G_j+I_{1j}+E_1+e_{1jk}\\ z_{2jl}=\mu+G_j+I_{2j}+E_2+e_{2jl}\\ \end{split} \tag{15.6} \]
Sin embargo, teniendo en cuenta de que se trata de solamente dos ambientes los que estamos comparando y como \(E_1=\mu_1-\mu\) y \(E_2=\mu_2-\mu\), al mismo tiempo que \(\mu=\frac{\mu_1+\mu_2}{2}\), entonces
\[ \begin{split} \mu_1=E_1+\mu \\ \mu_2=E_2+\mu \\ \\ \mu=\frac{E_1+\mu+E_2+\mu}{2} \Leftrightarrow \mu=\mu+\frac{E_1+E_2}{2} \Leftrightarrow \\ 0=\frac{E_1+E_2}{2} \Leftrightarrow \frac{E_1}{2}=-\frac{E_2}{2} \Leftrightarrow E_1=-E_2 \end{split} \tag{15.7} \]
por lo que vamos a reducir un parámetros en las ecuaciones del modelo (15.6) ya que \(E=E_1=-E_2\ \therefore\ E_2=-E\). Por otra parte, \(I_{1j}=\mu_{1j}-\mu-G_j-E\) y \(I_{2j}=\mu_{2j}-\mu-G_j+E\), además de que \(G_j=\frac{(\mu_{1j}+\mu_{2j})}{2}-\mu\). Usando este último resultado, tenemos que
\[ \begin{split} G_j=\frac{(\mu_{1j}+\mu_{2j})}{2} - \mu \Leftrightarrow 2\ (G_j+\mu)=\mu_{1j}+\mu_{2j} \Leftrightarrow \\ \mu_{1j}=2G_j+2\mu-\mu_{2j} \end{split} \tag{15.8} \]
y sustituyendo en \(I_{1j}=\mu_{1j}-\mu-G_j-E\) nos queda
\[ \begin{split} I_{1j}=2G_j+2\mu-\mu_{2j}-\mu-G_j-E=G_j+\mu-\mu_{2j}-E=\\ =-[\mu_{2j}-\mu-G_j+E]=-I_{2j} \end{split} \tag{15.9} \]
por lo que \(I_{1j}=-I_{2j}=I_j\). Utilizando los resultados de las ecuaciones (15.7) y (15.9) y sustituyendo en la ecuación (15.6) nos queda una versión más simplificada del modelo
\[ \begin{split} z_{1jk}=\mu+G_j+I_j+E+e_{1jk}\\ z_{2jl}=\mu+G_j-I_j-E+e_{2jl}\\ \end{split} \tag{15.10} \]
Las propiedades esenciales de este modelo lineal pueden resumirse de la siguiente manera:
La media total del modelo, es decir de todos los genotipos a través de todos los ambientes es igual a \(\mu\)
Los efectos genéticos de los grupos de individuos (familias) se asumen aleatorios, con esperanza de cero y varianza \(\sigma^2_G\)
De la misma manera, para un ambiente dado los efectos de interacción son variables aleatorias con esperanza de cero y varianza \(\sigma^2_I\)
Dentro de los grupos genéticos el efecto medio de interacción está restringido a que debe ser cero
Puede existir correlación entre los valores genotípicos y los efectos de interacción, en cuyo caso la covarianza es de \(\sigma_{G,I}\)
El valor promedio de los dos ambientes es cero
Las desviaciones residuales dentro de los ambiente tienen esperanza de cero y varianza \(\sigma^2_e\) para todos los grupos genéticos.
Dentro de cada ambiente, los efectos genotípicos son los directos del grupo genotípico y los de interacción, que como vimos antes estaban restringidos entre sí, por lo que \(G_{1j}=G_j+I_j\) y \(G_{2j}=G_j-I_j\).
Las varianzas genéticas de los grupos (familias) en los dos ambientes serán por lo tanto
\[ \begin{split} \sigma^2_G(1)=\sigma^2(G_j+I_j)=\sigma^2_G+\sigma^2_I+2 \sigma_{G,I}\\ \sigma^2_G(2)=\sigma^2(G_j-I_j)=\sigma^2_G+\sigma^2_I-2 \sigma_{G,I} \end{split} \tag{15.11} \]
mientras que para la covarianza genética de los grupos entre los dos ambientes tenemos
\[ \begin{split} \sigma_G(1,2)=\sigma[(G_j+I_j),(G_j-I_j)]=\sigma(G_j,G_j)+\sigma(G_j,I_j)-\sigma(G_j,I_j)+\sigma(I_j,-I_j)=\\ \sigma(G_j,G_j)-\sigma(I_j,I_j)=\sigma^2_G-\sigma^2_I \end{split} \tag{15.12} \]
Por otra parte, como vimos en la sección Semejanza entre parientes del capítulo Parentesco y Semejanza entre Parientes, la varianza entre familias es igual a la covarianza fenotípica entre individuos de la misma familia. Por lo tanto, la contribución genética aditiva a la varianza genética en los ambientes 1 y 2, es decir a \(\sigma^2_G(1)\) y \(\sigma^2_G(2)\) será igual a \(2\Theta\sigma^2_A(1)=a_{XY}\sigma^2_A(1)\) y \(2\Theta\sigma^2_A(2)=a_{XY}\sigma^2_A(2)\), mientras que el aporte de la covarianza genética aditiva a la covarianza genética aditiva \(\sigma_G(1,2)\) será \(2\Theta\sigma_A(1,2)=a_{XY}\sigma_A(1,2)\). \(\Theta\) es el coeficiente de coancestría entre los miembros del grupo familiar, idéntico a lo que en la sección El concepto de “identidad por ascendencia” (IBD) manejamos como \(f_{XY}\), mientras que \(a_{XY}\) es el parentesco aditivo (que en ese contexto notamos con \(r_{XY}\)); recordar que \(\Theta=\frac{1}{2}a_{XY}\) o lo que es lo mismo \(f_{XY}=\frac{1}{2}r_{XY}\).
A partir de las varianzas y covarianza genético-aditiva, ignorando los efectos genéticos no-aditivos, podemos definir el coeficiente de correlación genética (aditiva) entre ambientes como
\[ \begin{split} \rho_x=\frac{\sigma_A(1,2)}{\sigma_A(1)\sigma_A(2)}=\frac{2\Theta\ \sigma_A(1,2)}{\sqrt{2\Theta\ \sigma^2_A(1)\ 2\Theta\ \sigma^2_A(2)}}=\frac{\sigma_A(1,2)}{\sigma_A(1)\ \sigma_A(2)}=\frac{\sigma_G(1,2)}{\sigma_G(1)\ \sigma_G(2)} \end{split} \tag{15.13} \]
La importancia de la ecuación (15.13) radica en que a partir de estimaciones de las varianzas (\(\sigma^2_G(1)\) y \(\sigma^2_G(2)\) ) y covarianzas (\(\sigma_G(1,2)\) ) de familias podemos estimar la correlación genética de la característica entre ambientes diferentes.
En las ecuaciones anteriores trabajamos con las varianzas y covarianzas de las medias familiares de grupos genotípicos (familias), que tienen componentes de varianza genotípica y de interacción. Ahora, operando con las ecuaciones (15.11) y (15.12) tenemos que podemos obtener la varianza genética como
\[\frac{1}{2}[\frac{\sigma^2_G(1)+\sigma^2_G(2)}{2}+\sigma_G(1,2)]=\] \[\frac{1}{2}[\frac{\sigma^2_G+\sigma^2_I+2 \sigma_{G,I}+\sigma^2_G+\sigma^2_I-2 \sigma_{G,I}}{2}+\sigma^2_G-\sigma^2_I]=\] \[\frac{1}{2}[\frac{2\sigma^2_G+2\sigma^2_I}{2}+\sigma^2_G-\sigma^2_I]=\sigma^2_G\ \therefore\ \] \[ \begin{split} \sigma^2_G=\frac{1}{2}[\frac{\sigma^2_G(1)+\sigma^2_G(2)}{2}+\sigma_G(1,2)] \end{split} \tag{15.14} \]
mientras que la varianza de la interacción la podemos obtener de la siguiente forma
\[\frac{1}{2}[\frac{\sigma^2_G(1)+\sigma^2_G(2)}{2}-\sigma_G(1,2)]=\] \[=\frac{1}{2}[\frac{\sigma^2_G+\sigma^2_I+2 \sigma_{G,I}+\sigma^2_G+\sigma^2_I-2 \sigma_{G,I}}{2}-\sigma^2_G+\sigma^2_I]=\] \[=\frac{1}{2}[\frac{2\sigma^2_G+2\sigma^2_I}{2}-\sigma^2_G+\sigma^2_I]=\sigma^2_I\ \therefore\ \] \[ \begin{split} \sigma^2_I=\frac{1}{2}[\frac{\sigma^2_G(1)+\sigma^2_G(2)}{2}-\sigma_G(1,2)] \end{split} \tag{15.15} \]
De la misma manera, la covarianza entre los efectos genéticos y de interacción se puede obtener a partir de
\[\frac{\sigma^2_G(1)-\sigma^2_G(2)}{4}=\frac{\sigma^2_G+\sigma^2_I+2 \sigma_{G,I}-\sigma^2_G-\sigma^2_I+2 \sigma_{G,I}}{4}=\frac{4 \sigma_{G,I}}{4}= \sigma_{G,I}\ \therefore\ \] \[ \begin{split} \sigma_{G,I}=\frac{\sigma^2_G(1)-\sigma^2_G(2)}{4} \end{split} \tag{15.16} \]
como \((\sigma^2_G+\sigma^2_I)^2=\sigma^4_G+\sigma^4_I+2 \sigma^2_G \sigma^2_I\) y \((\sigma^2_G+\sigma^2_I+2\sigma_{G,I})(\sigma^2_G+\sigma^2_I-2\sigma_{G,I})=\sigma^4_G+\sigma^4_I+2 \sigma^2_G \sigma^2_I-4\sigma^2_{G,I}\), el producto de \((\sigma^2_G+\sigma^2_I+2\sigma_{G,I})(\sigma^2_G+\sigma^2_I-2\sigma_{G,I})\) puede escribirse como \((\sigma^2_G+\sigma^2_I)^2-4\sigma^2_{G,I}\) y entonces
\[ \begin{split} \rho_x=\frac{\sigma^2_G-\sigma^2_I}{\sqrt{(\sigma^2_G+\sigma^2_I)^2-4\sigma^2_{G,I}}} \end{split} \tag{15.17} \]
El análisis de la ecuación (15.17) nos permite entender mejor el comportamiento de la característica de interés en dos ambientes, a la luz de la posible interación genotipo-ambiente. En primer lugar, solo como corroboración, en caso de que la interacción genotipo-ambiente sea nula, entonces \(\sigma^2_I=\sigma_{G,I}=0\) y por lo tanto
\[ \begin{split} \rho_x=\frac{\sigma^2_G-0}{\sqrt{(\sigma^2_G+0)^2-4 \times 0}}=\frac{\sigma^2_G}{\sqrt{\sigma^4}}=\frac{\sigma^2_G}{\sigma^2_G}=1 \end{split} \]
o lo que es lo mismo, la correlación genética entre ambientes es igual a uno, es decir, se trata de la misma característica (pese a haberla considerado como dos características diferentes para el análisis).
Por otra parte, si existe una correlación negativa perfecta entre los rankings de los grupos genéticos (familias) a través de los ambientes, entonces la varianza genética de los grupos es cero, es decir \(\sigma^2_G=0\) y por lo tanto \(\sigma_{G,I}=0\) (ya que es la covarianza de una variable aleatoria con una constante). Colocando esos valores en la ecuación (15.17), tenemos que en ese caso, la correlación genética entre ambientes es
\[ \begin{split} \rho_x=\frac{0-\sigma^2_I}{\sqrt{(0+\sigma^2_I)^2-4 \times 0}}=-\frac{\sigma^2_I}{\sigma^2_I}=-1 \end{split} \]
Finalmente, cuando las varianzas entre familias en los dos ambientes son iguales, de acuerdo a la ecuación (15.11), \(\sigma^2_G+\sigma^2_I+2 \sigma_{G,I}=\sigma^2_G+\sigma^2_I-2 \sigma_{G,I} \Leftrightarrow \sigma_{G,I}=0\). Por lo tanto, de acuerdo con la ecuación (15.17), sustituyendo ese valor tenemos
\[ \begin{split} \rho_x=\frac{\sigma^2_G-\sigma^2_I}{\sqrt{(\sigma^2_G+\sigma^2_I)^2-4 \times 0}}=\frac{\sigma^2_G-\sigma^2_I}{\sqrt{(\sigma^2_G+\sigma^2_I)^2}}=\frac{\sigma^2_G-\sigma^2_I}{\sigma^2_G+\sigma^2_I} \end{split} \]
Claramente, de acuerdo con esta última ecuación, si las varianzas entre familias en los dos ambientes son iguales, entonces (como las varianzas deben ser positivas) la presencia de interacción disminuye el valor de la correlación genética por debajo de 1.
PARA RECORDAR
Podemos describir los fenotipos de dos individuos \(k\) y \(l\), ambos pertenecientes al mismo grupo genotípico \(j\), para la característica de interés en los dos ambientes, \(1\) y \(2\) como: \(z_{1jk}=\mu+G_j+I_j+E+e_{1jk}\\z_{2jl}=\mu+G_j-I_j-E+e_{2jl}\)
Las propiedades esenciales de este modelo lineal pueden resumirse en: 1) la media total del modelo, es decir de todos los genotipos a través de todos los ambientes es igual a \(\mu\), 2) los efectos genéticos de los grupos de individuos (familias) se asumen aleatorios, con esperanza de cero y varianza \(\sigma^2_G\), 3) de la misma manera, para un ambiente dado los efectos de interacción son variables aleatorias con esperanza de cero y varianza \(\sigma^2_I\), 4) dentro de los grupos genéticos el efecto medio de interacción está restringido a que debe ser cero, 5) puede existir correlación entre los valores genotípicos y los efectos de interacción, en cuyo caso la covarianza es de \(\sigma_{G,I}\), 6) el valor promedio de los dos ambientes es cero, 7) las desviaciones residuales dentro de los ambiente tienen esperanza de cero y varianza \(\sigma^2_e\) para todos los grupos genéticos.
Las varianzas genéticas de los grupos (familias) en los dos ambientes serán \(\sigma^2_G(1)=\sigma^2(G_j+I_j)=\sigma^2_G+\sigma^2_I+2 \sigma_{G,I}\\\sigma^2_G(2)=\sigma^2(G_j-I_j)=\sigma^2_G+\sigma^2_I-2 \sigma_{G,I}\)
La covarianza genética de los grupos entre los dos ambientes será: \(\sigma_G(1,2)=\sigma^2_G-\sigma^2_I\)
A su vez, el coeficiente de correlación genética (aditiva) entre ambientes quedó definido como: \(\rho_x=\frac{\sigma_G(1,2)}{\sigma_G(1)\ \sigma_G(2)}\).
Entonces, a partir de estimaciones de las varianzas (\(\sigma^2_G(1)\) y \(\sigma^2_G(2)\) ) y covarianzas (\(\sigma_G(1,2)\) ) de familias podemos estimar la correlación genética de la característica entre ambientes diferentes.
En caso de que la interacción genotipo-ambiente sea nula, entonces \(\sigma^2_I=\sigma_{G,I}=0\) y por lo tanto la correlación genética entre ambientes es igual a uno, es decir, se trata de la misma característica (pese a haberla considerado como dos características diferentes para el análisis). Análogamente, si existe una correlación negativa perfecta entre los rankings de los grupos genéticos (familias) a través de los ambientes, entonces la varianza genética de los grupos es cero, es decir \(\sigma^2_G=0\) y por lo tanto \(\sigma_{G,I}=0\) (ya que es la covarianza de una variable aleatoria con una constante). Por lo que en ese caso, la correlación genética entre ambientes es -1.
Procedimientos de estimación
A partir de los resultados obtenidos en la sección anterior resulta claro que la determinación de la existencia de interacción entre genotipo y ambiente a partir del método de la correlación consiste en estimar su valor y determinar si el mismo es significativamente menor a uno. Es decir, un valor de \(\rho_x=1\) es indicación de que lo que consideramos como dos características desde el punto de vista genético es en realidad la misma y que por lo tanto no existe interacción GxE. El problema es que aún siendo la correlación igual a uno, el valor estimado es una variable aleatoria, con su propia distribución y que posiblemente casi siempre nos dé un valor estimado menor a uno, aún siendo uno su valor verdadero. Por lo tanto, tenemos dos problemas diferentes, uno de estimación y el otro de significación estadística.
Para obtener un estimado \(r_x\) de \(\rho_x\), el procedimiento más sencillo consiste en separar la familias en dos y ensayar cada mitad en uno de los ambientes. Eso nos proporcionará estimados de las varianzas entre familias en los dos ambientes (\(\sigma^2_G(1)\) y \(\sigma^2_G(2)\)), así como un estimado de la covarianza de familia entre ambientes (\(\sigma_G(1,2)\)). A partir de esto, alcanza con sustituir los valores observados por los valores esperados en la ecuación (15.13), es decir
\[ \begin{split} \rho_x=\frac{\sigma_G(1,2)}{\sigma_G^2(1)\ \sigma_G^2(2)} \end{split} \]
El procedimientos más sencillo para obtener estimados de \(\sigma^2_G(1)\) y \(\sigma^2_G(2)\) es a través de un análisis de varianza, como el que hemos visto en capítulos precedentes, mientras que para la estimación de la covarianza en procedimiento más sencillo parece ser el cálculo de la covarianza observada entre medias familiares en los dos ambientes. Una advertencia importante aquí es la de evitar el uso de estructuras familiares en las que se compartan otros factores, por ejemplo los efectos maternales ya que estos contaminarán las estimaciones.
Como la estimación de las varianzas y covarianzas son obtenidas indirectamente, las correlación obtenida por este método no se trata de una correlación producto-momento (la correlación clásica, o de Pearson) y por lo tanto las estimaciones pueden escaparse del intervalo natural (\(\pm 1\)). Como Michael Lynch and Walsh (1998) sugieren, una forma sencilla de obtener una correlación producto-momento es a través de la regresión de las medias familiares en los dos ambientes, por ejemplo \(\bar z_{1j}\) en \(\bar z_{2j}\).
Otra alternativa usada en la de obtener \(r_x\) directamente a partir de la correlación de las medias familiares \(r(\bar z_{1j},\bar z_{2j})\) como un estimador de la correlación genética entre ambientes. Sin embargo, como estos autores remarcan, la correlación estimada de esta forma se encuentra sesgada hacia abajo, ya que en el denominador de la misma aparecen los efectos del error de medición y del ambiente inflando las correspondientes varianzas. Esto lleva a una tendencia a declarar la existencia de interacción genotipo-ambiente ya que puede sugerir valores significativamente menores a uno, aún cuando la correlación genética sea de uno.
Cuando existen datos disponibles para más de dos ambientes una alternativa es el uso de un análisis de varianza de dos vías, siendo los dos factores los genotipos (familias) y los ambientes. Esto nos introduce en un tema de modelos con efectos aleatorios y una mezcla de efectos aleatorios y fijos, cuya interpretación es algo diferente entre ellos. Si consideramos que los genotipos son una muestra aleatoria de una distribución de genotipos posibles (por ejemplo, de “familias”), entonces los efectos macroambientales pueden ser considerados de dos maneras diferentes, como aleatorios o como fijos. En el primer caso se trataría de que los ambientes también son una muestra representativa de los ambientes posibles, como cuando elegimos al azar diferentes tambos, o regiones (macroambientes) para ensayar los genotipos, o cuando elegimos al azar parcelas donde probar variedades de maíz. En este caso, tenemos un modelo aleatorio, ya que tanto los genotipos como los macroambientes son ambos aleatorios. En el segundo caso, si consideramos una serie particular de ambientes de interés, por ejemplo diferentes países o regiones donde evaluar los genotipos, que son las únicas de interés, entonces vamos a tratar con modelos mixtos, ya que los genotipos serán aleatorios, mientras que los macroambientes serán considerados efectos fijos. Para entender las diferencias entre ambos tipos de modelos se recomienda la lectura del capítulo 22 de Michael Lynch and Walsh (1998), en especial la sección “TWO-WAY ANALYSIS OF VARIANCE”.
El uso de modelos mixtos para el estudio de la interacción genotipo ambiente a partir de datos existentes en las evaluaciones genéticas nacionales e internacionales ha sido extenso. Ejemplos locales y regionales incluyen los estudios realizados por H. Naya, Urioste, and Franco (2002) en vacas lecheras a partir de la definición de “clusters ambientales” obtenidos de los propios datos de la evaluación; el trabajo de Espasandin et al. (2013), comparando las poblaciones Angus de Uruguay y Brasil o el trabajo de Zwald et al. (2003), investigando los factores que afectan el resultado de vacas Holstein en 17 países que participaban de la evaluación MACE realizada por INTERBULL.
15.4 Genética cuantitativa de la interacción GxE
En todo el tratamiento que dimos previamente a la genética cuantitativa asumimos que genotipo era sinónimo de la acción de los genes sobre el fenotipo de los individuos y que podíamos aislar ese concepto del concepto de ambiente. Sin embargo, como lo muestra Jong (1990), en el trabajo original de Woltereck (1909) donde aparece por primera vez el concepto de norma de reacción, el concepto del mismo Woltereck era de que ‘Der “Genotypus” (die genotyp. Grundlage) eines Quantitativmerkmals ist die vererbte Reaktionsnorm’ (‘El “genotipo” (la base genotípica) de un rasgo cuantitativo es la norma de reacción heredada’), a tal punto que el encabezado de la sección luce “Genotypus = Reaktionsnorm”. Es decir, en la concepción original de Woltereck aparece que no podemos separar genotipo de ambiente ya que para él, el concepto de genotipo es precisamente la forma específica de reaccionar a los ambientes que tienen los individuos con base genética.
En este capítulo hemos ido avanzando en la comprensión de la importancia que tiene la interacción entre el genotipo y el ambiente, pero apenas nos hemos asomado en el impacto que tiene la misma en los principales resultados que hemos derivado para la genética cuantitativa. De acuerdo con Michael Lynch and Walsh (1998), lamentablemente pocos avances se han hecho en la materia, aunque el trabajo de Jong (1990) sugiere un camino simple e interesante, por lo que vamos a delinear sus principios aquí como forma de conectar el contenido de este capítulo con los capítulos previos.
Supongamos que tenemos una característica fenotípica que se encuentra gobernada por un gen con dos alelos, con frecuencias \(p\) y \(q\) como usualmente. La única diferencia en nuestro planteo actual respecto a lo que vimos antes en el capítulo El modelo genético básico es que ahora el Efecto medio de los alelos ya no es independiente del ambiente sino que es una función lineal del mismo. Por lo tanto, con esta diferencia, los efectos medios de los alelos serán ahora del tipo
\[ \begin{split} \alpha_1=a_1+c_1 E\\ \alpha_2=a_2+c_2 E\\ \end{split} \tag{15.18} \]
con \(a_1\) y \(a_2\) los interceptos y \(c_1\) y \(c_2\) las pendientes de la regresión del valor medio del alelo en el gradiente ambiental.
Como la respuesta genotípica será ahora la norma de reacción, entonces, para una combinación específica de alelos \(i\) y \(j\), la misma será
\[ \begin{split} R_{ij}=\alpha_i+\alpha_j=(a_i+c_i E)+(a_j+c_j E)\ \therefore\ \\ R_{ij}=(a_i+a_j)+(c_i+c_j) E \end{split} \tag{15.19} \]
En características determinadas por varios genes de acción aditiva entre ellos, la norma de reacción será igual a la suma de los \(R_{ij}\) de todos los loci involucrados, cada uno con sus frecuencias alélicas y funciones lineales de \(E\).
En el capítulo El Modelo Genético Básico vimos que el efecto de sustitución era igual a \(\alpha=\alpha_1-\alpha_2\) y que de acuerdo a la ecuación (8.44) la varianza aditiva era igual a \(\sigma^2_A=2pq\alpha^2\). Por lo tanto, el efecto de sustitución será \(\alpha=(a_1+c_1 E)-(a_2+c_2E)=(a_1-a_2)+(c_1-c_2)E\) y la varianza aditiva será
\[ \begin{split} \sigma^2_A=2pq[(a_1-a_2)+(c_1-c_2)E]^2 \end{split} \tag{15.20} \]
La expansión de la ecuación anterior nos lleva a
\[ \begin{split} \sigma^2_A=2pq[(a_1-a_2)^2+2E(a_1-a_2)(c_1-c_2)+(c_1-c_2)^2E^2]=\\ =2pq(a_1-a_2)^2+2pq[2E(a_1-a_2)(c_1-c_2)]+2pq(c_1-c_2)^2E^2 \end{split} \tag{15.21} \]
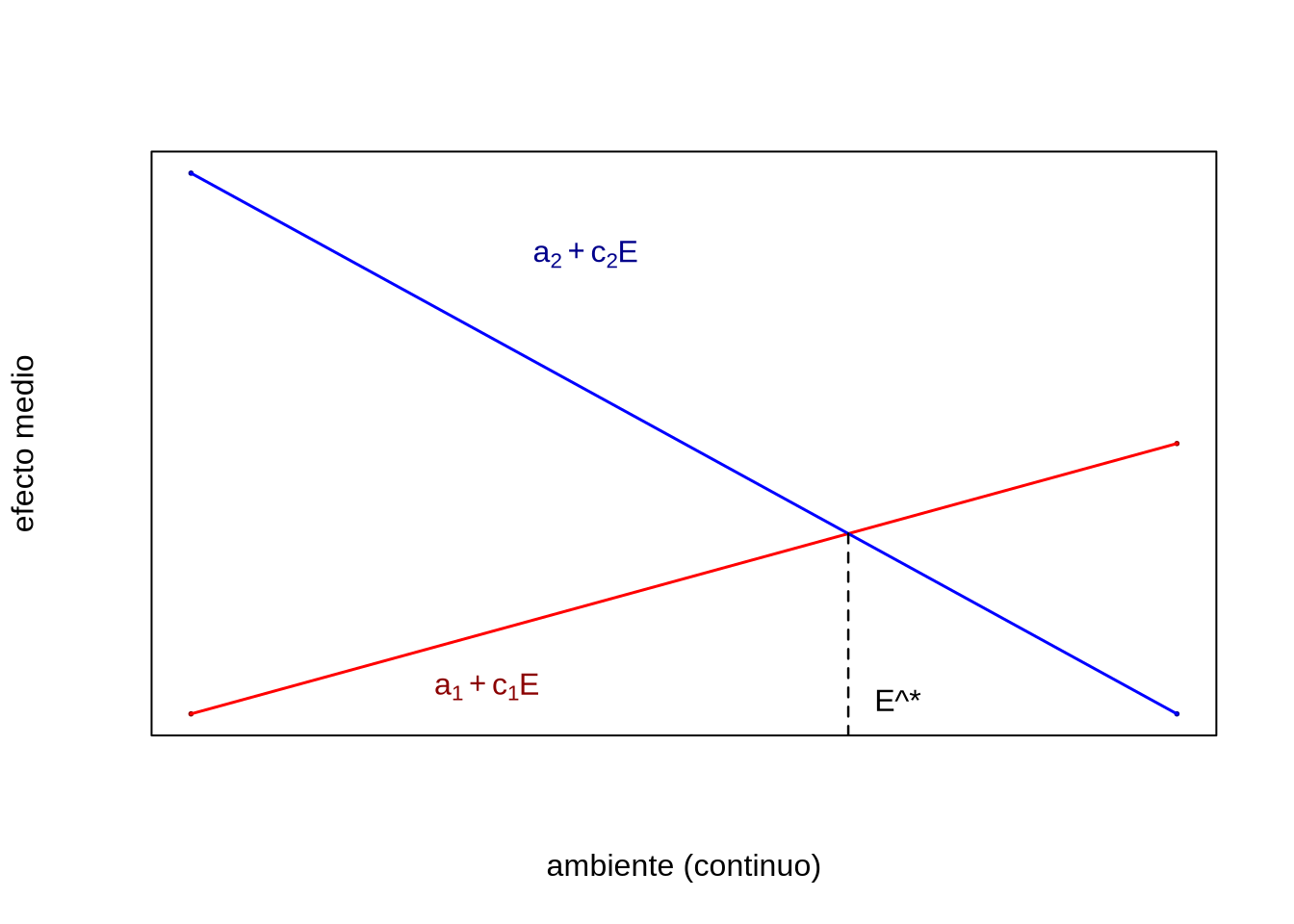
Figura 15.6: Normas de reacción lineales para el efecto medio de un alelo. El punto \(E^{\star}\) representa el valor del macroambiente donde se produce intersección de las dos funciones lineales (es decir, los efectos medios son iguales y el efecto de sustitución igual a cero).
Considerando un punto determinado del macroambiente \(E\), es decir, considerado como una constante y reconociendo cada uno de los términos de la ecuación (15.21) como una varianza o covarianza, podemos expresar la varianza aditiva bajo la norma de reacción como
\[ \begin{split} \sigma^2_A=\sigma^2_a+2E\sigma_{a,c}+E^2\sigma^2_c \end{split} \tag{15.22} \]
es decir, la suma de las varianzas y covarianza de interceptos y pendientes que forman la norma de reacción.
Por lo tanto, de acuerdo a la ecuación (15.22), la varianza aditiva de la característica no será más un valor solamente dependiente de \(\alpha\) y de las frecuencias, sino que ahora será una función del macroambiente. De hecho, mirando la función \(\sigma^2_A(E)\) apreciamos que se trata de una función parabólica. Como además las varianzas deben ser positivas, el coeficiente del término cuadrático (\(\sigma^2_c\)) en la función es positivo y por lo tanto se trata de una parábola cóncava (con los brazos extendidos hacia arriba). El mínimo de la función estará dado además por el valor en que la derivada se hace cero, que es
\[ \begin{split} \frac{d\sigma^2_A(E)}{dE}=\frac{d[\sigma^2_a+2E\sigma_{a,c}+E^2\sigma^2_c]}{dE}=2\sigma_{a,c}+2\sigma^2_cE\ \therefore\ \\ 2\sigma_{a,c}+2\sigma^2_cE=0 \Leftrightarrow E=-\frac{\sigma_{a,c}}{\sigma^2_c}=-\frac{(a_1-a_2)(c_1-c_2)}{(c_1-c_2)^2} \\ E = -\frac{(a_1-a_2)}{(c_1-c_2)} \end{split} \]
En la Figura 15.7 se representa la superficie correspondiente a la varianza aditiva, tal cual aparece en la ecuación (15.20). La misma es función de la frecuencia de uno de los dos alelos, \(p\) (ya que \(q=1-p\)) y del macroambiente \(E\) ya que en este caso las normas de reacción de los genotipos son lineales y no paralelas. Como es de esperar, la varianza tiene forma parabólica convexa con respecto a \(p\) y con máximo en \(p=q=\frac{1}{2}\), mientras que también tiene forma parabólica, pero cónica con respecto al ambiente y con mínimo en \(E^{\star}\).
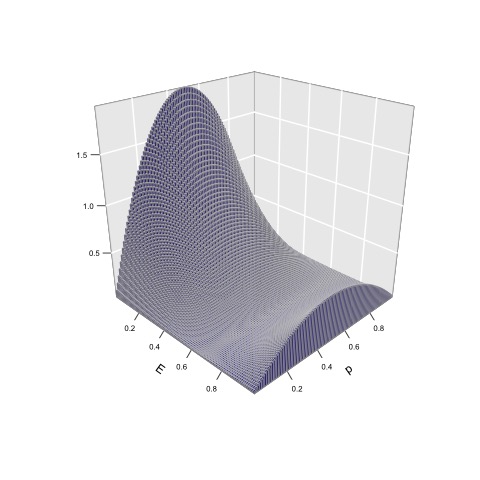
Figura 15.7: Superficie de la varianza aditiva para normas de reacción lineales en función de la frecuencia \(p\) de uno de los dos alelos y del valor del macroambiente (arbitrario, representado entre cero y uno).
Una forma complementaria de entender las consecuencias de esta dependencia de la varianza aditiva en el valor del macroambiente es considerar que como \(\alpha_1\) y \(\alpha_2\) son funciones lineales de \(E\), existen dos posibilidades para las rectas que estas ecuaciones describen: a) son parelelas, o b) se cortan. Si son paralelas, entonces las pendientes \(c_1\) y \(c_2\) son iguales, por lo que \(c_1-c_2=0\) y la ecuación (15.20) se transforma en
\[ \begin{split} \sigma^2_A=2pq[(a_1-a_2)+(c_1-c_2)E]^2=2pq(a_1-a_2)^2 \end{split} \]
y no hay dependencia de la varianza aditiva en el ambiente, es decir no se observa un efecto de interacción genotipo ambiente. Este resultado también podría obtenerse de la ecuación (15.22), ya que si \(c_1=c_2\) entonces \(\sigma^2_c=\sigma_{a,c}=0\) (ya que \(c\) es constante).
En el caso de que \(\alpha_1\) y \(\alpha_2\) no sean paralelas, entonces las mismas deben cortarse en algún punto. Si igualamos ambas funciones tenemos que
\[ \begin{split} \alpha_1=\alpha_2 \Leftrightarrow a_1+c_1E=a_2+c_2E \Leftrightarrow E(c_1-c_2)=(a_2-a_1)\ \therefore\ \\ E^{\star}=\frac{(a_1-a_2)}{(c_2-c_1)} \end{split} \tag{15.23} \]
donde \(E^{\star}\) es el ambiente correspondiente al punto de corte de las funciones de efecto medio. Si sustituimos dicho valor en la función de la varianza aditiva dada por la ecuación (15.20) tenemos que
\[\sigma^2_A=2pq[(a_1-a_2)+(c_1-c_2)E]^2=2pq[(a_1-a_2)+(c_1-c_2)\frac{(a_1-a_2)}{(c_2-c_1)}]^2=\] \[2pq[(a_1-a_2)-(a_1-a_2)]^2=2pq [0]^2\ \therefore\ \] \[ \begin{split} \sigma^2_A=0 \end{split} \tag{15.24} \]
Por lo tanto, si tenemos que las normas de reacción son lineales y existe un efecto de interacción, entonce encontramos un valor ambiental en el que la varianza aditiva de la característica es igual a cero, por lo que no responderá a la selección (la heredabilidad de la característica es igual a cero). Como para cada locus los interceptos y pendientes son específicos, también lo serán los puntos de corte y por lo tanto para características poligénicas resulta difícil que en algún punto la varianza aditiva sea cero. Sin embargo, la posibilidad es firme de que exista uno o más puntos del macroambiente en que la varianza aditiva se vea reducida sustancialmente.
Un punto final al que este análisis nos permite arribar fácilmente es al del estudio de la correlación genética de la característica expresada en dos ambientes, \(E_j\) y \(E_k\). En este caso, de forma análoga a lo derivado para la varianza aditiva en la ecuación (15.20), la covarianza genética estará dada por
\[ \begin{split} \sigma_{A_{j},A_{k}}=2pq[(a_1-a_2)+(c_1-c_2)E_j][(a_1-a_2)+(c_1-c_2)E_k] \end{split} \tag{15.25} \]
De acuerdo con esta última ecuación, si cualquiera de los dos ambientes es igual al punto de corte de los efectos medios, es decir \(E_j=E^{\star}\) o \(E_k=E^{\star}\), entonces la covarianza genética de la característica será igual a cero y lo mismo para la correlación genética. Por otra parte, si \(E_j\) y \(E_k\) se encuentran ambos del mimso lado respecto a \(E^{\star}\) (a la izquierda o a la derecha de dicho punto), entonces el producto \([(a_1-a_2)+(c_1-c_2)E_j][(a_1-a_2)+(c_1-c_2)E_k]\) será positivo y la correlación genética será también positiva. Sin embargo, si los dos puntos se encuentran en lados diferentes respecto a \(E^{\star}\), entonces el producto será negativo y también lo será la correlación genética. Si bien se trata de un modelo teórico elemental, el mismo permite explicar correlaciones genéticas negativas de la misma característica cuando la misma se expresa en dos ambientes diferentes y es un insumo importante para construir modelos más complejos.
Ejemplo 15.2
Una determinada característica fenotípica se encuentra determinada por un locus con dos alelos. Para esta característica existe interacción entre genotipo y ambiente ya que los efectos medios de los dos alelos son \(\alpha_1=6-E\) y \(\alpha_2=4+2E\), siendo \(E\) la variable (continua) ambiental que determina el valor de los alelos.
Graficar los efectos medios de los genes en función del ambiente (entre \(E=0\) y \(E=1\)) y calcular el punto en que son iguales. Determinar la varianza aditiva de la característica como función del ambiente \(E\) y graficarla entre los valores de \(E=0\) y \(E=1\) para una frecuencia del alelo \(A_1\) \(p=0,25\) y para \(p=0,50\).
La Figura 15.8 representa los efectos medios de ambos alelos como función del ambiente \(E\), entre los valores de \(E=0\) y \(E=1\). De acuerdo a la ecuación (15.23), el punto de corte se va a encontrar en
\[ \begin{split} E^{\star}=\frac{(a_1-a_2)}{(c_2-c_1)}=\frac{6-4}{2-(-1)}=\frac{2}{3}=0,666 \end{split} \]
lo que aparece marcado en la figura.
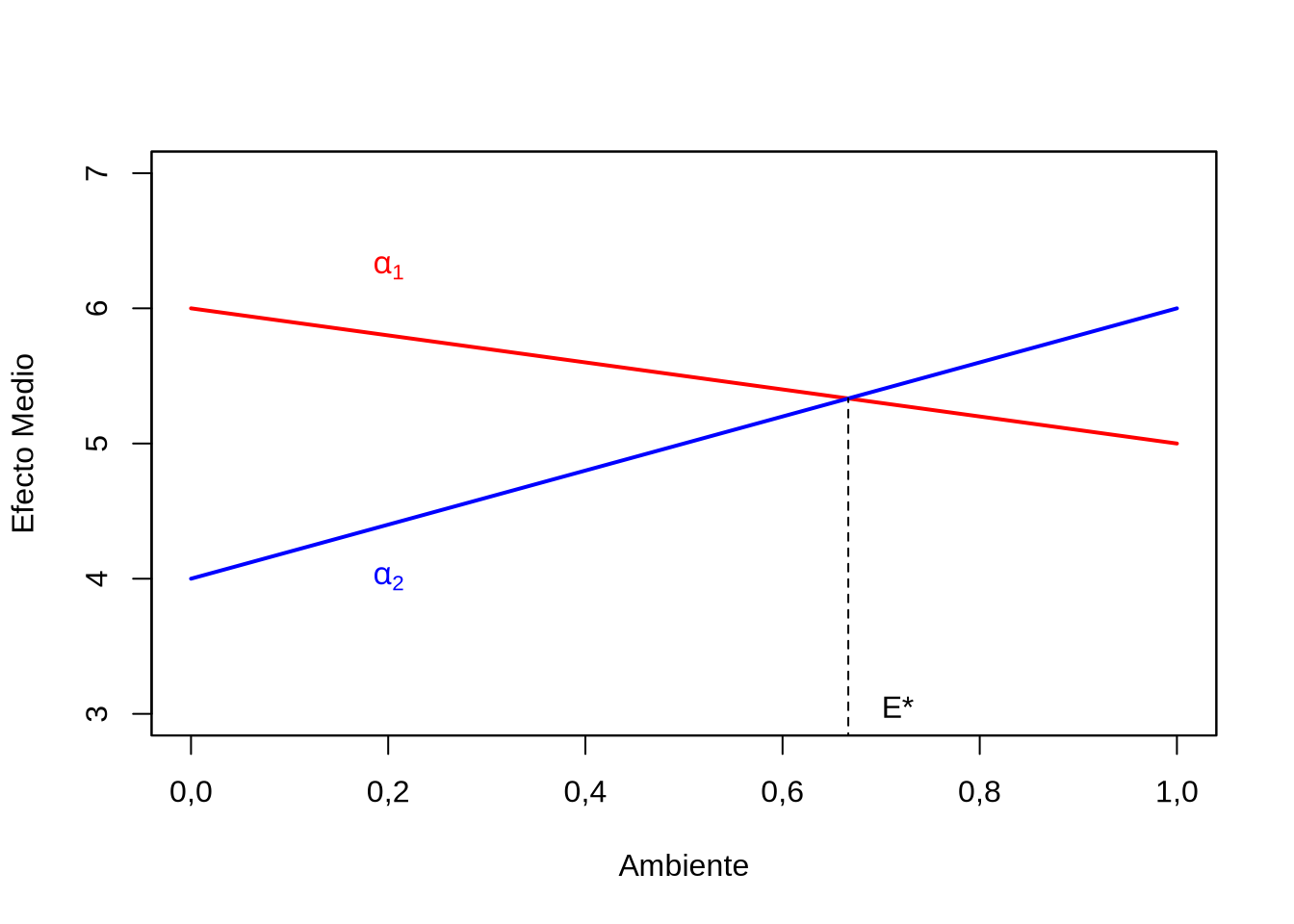
Figura 15.8: Efectos medios de los dos alelos \(\alpha_1=6-E\) y \(\alpha_2=4+2E\), como función del ambiente \(E\). El punto de corte aparece marcado por \(E^{\star}\).
La varianza aditiva es una función del ambiente. De acuerdo a la ecuación (15.20) la misma es igual a
\[ \begin{split} \sigma^2_A=2pq[(a_1-a_2)+(c_1-c_2)E]^2=2pq[(6-4)+(-1-2)E]^2=2pq[2-3E]^2 \end{split} \]
Sustituyendo ahora el valor de \(p=0,25\) nos queda
\[ \begin{split} \sigma^2_A=2pq[2-3E]^2=2\times 0,25 \times 0,75[2-3E]^2=0,375[2-3E]^2 \end{split} \]
mientras que para \(p=0,5\) nos queda
\[ \begin{split} \sigma^2_A=2pq[2-3E]^2=2\times 0,5 \times 0,5[2-3E]^2=0,5[2-3E]^2 \end{split} \]
La representación gráfica de estas funciones de varianza aparece en la Figura 15.9, donde se aprecia claramente la forma parabólica de la varianza aditiva, con un mínimo en \(E=\frac{2}{3}=E^{\star}\), ya que en ese valor \(\sigma^2_A=2pq[2-3(\frac{2}{3})]^2=0\).
![Variamnza aditiva de la característisca en función del ambiente \(E\), para dos frecuencias alélicas del alelo \(A_1\). En rojo \(p=0,25\), en azul \(p=0,5\). Como la función de varianza aditiva es igual a \(\sigma^2_A=2pq[2-3E]^2\), cuando \(E=\frac{2}{3}=E^{\star}\) la varianza se hace cero.](ApuntesGeneticaII_files/figure-html/figura15p201-1.png)
Figura 15.9: Variamnza aditiva de la característisca en función del ambiente \(E\), para dos frecuencias alélicas del alelo \(A_1\). En rojo \(p=0,25\), en azul \(p=0,5\). Como la función de varianza aditiva es igual a \(\sigma^2_A=2pq[2-3E]^2\), cuando \(E=\frac{2}{3}=E^{\star}\) la varianza se hace cero.
PARA RECORDAR
Para obtener un estimado \(r_x\) de \(\rho_x\), el procedimiento más sencillo consiste en separar la familias en dos y ensayar cada mitad en uno de los ambientes. Eso nos proporcionará estimados de las varianzas entre familias en los dos ambientes (\(\sigma^2_G(1)\) y \(\sigma^2_G(2)\)), así como un estimado de la covarianza de familia entre ambientes (\(\sigma_G(1,2)\)).
Si tenemos una característica fenotípica que se encuentra gobernada por un gen con dos alelos, con frecuencias \(p\) y \(q\), el efecto medio de los alelos ya no es independiente del ambiente sino que es una función lineal del mismo, por lo que ahora serán: \(\alpha_1=a_1+c_1 E\\\alpha_2=a_2+c_2 E\\\) con \(a_1\) y \(a_2\) los interceptos y \(c_1\) y \(c_2\) las pendientes de la regresión del valor medio del alelo en el gradiente ambiental.
Como la respuesta genotípica será ahora la norma de reacción, entonces, para una combinación específica de alelos \(i\) y \(j\), la misma será \(R_{ij}=(a_i+a_j)+(c_i+c_j) E\). En características determinadas por varios genes de acción aditiva entre ellos, la norma de reacción será igual a la suma de los \(R_{ij}\) de todos los loci involucrados, cada uno con sus frecuencias alélicas y funciones lineales de \(E\).
La varianza aditiva bajo la norma de reacción será \(\sigma^2_A=\sigma^2_a+2E\sigma_{a,c}+E^2\sigma^2_c\), es decir, la suma de las varianzas y covarianza de interceptos y pendientes que forman la norma de reacción.
Dada la correlación genética de la característica expresada en dos ambientes, \(E_j\) y \(E_k\), la covarianza genética estará dada por: \(\sigma_{A_{j},A_{k}}=2pq[(a_1-a_2)+(c_1-c_2)E_j][(a_1-a_2)+(c_1-c_2)E_k]\). Por lo que si cualquiera de los dos ambientes es igual al punto de corte de los efectos medios, es decir \(E_j=E^{\star}\) o \(E_k=E^{\star}\), entonces la covarianza genética de la característica será igual a cero y lo mismo para la correlación genética. Por otra parte, si \(E_j\) y \(E_k\) se encuentran ambos del mimso lado respecto a \(E^{\star}\) (a la izquierda o a la derecha de dicho punto), entonces el producto \([(a_1-a_2)+(c_1-c_2)E_j][(a_1-a_2)+(c_1-c_2)E_k]\) será positivo y la correlación genética será también positiva. Sin embargo, si los dos puntos se encuentran en lados diferentes respecto a \(E^{\star}\), entonces el producto será negativo y también lo será la correlación genética.
15.5 Otros ejemplos de interacción genotipo-ambiente
En 1982, Gupta & Lewontin (Gupta and Lewontin (1982)) realizaron un estudio sobre normas de reacción en la mosca Drosophila pseudoobscura, variando la densidad de huevos y la temperatura y midiendo el número de setas abdominales. Para ello usaron 32 cepas isocromosómicas para el segundo cromosma. En la Figura 15.10 se ilustra alguno de los resultados obtenidos. Si bien los efectos medios del genotipo, el efecto de la temperatura y de la densidad fueron significativos, también aparecieron importantes efectos de interacción genotipo-ambiente, que en la figura aparecen como la falta de paralelismo en las líneas. Por otra parte, los autores observaron que alrededor del \(30-45\%\) de las comparaciones por pares entre genotipos mostraron un cambio en la posición relativa de los mismos al cambiar la temperatura. Por lo tanto, no es posible caracterizar un genotipo como si tuviera mayor número de setas que otro, ya que esto sólo puede ser determinado en forma relativo a un ambiente particular.
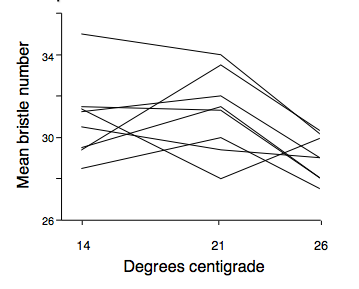
Figura 15.10: Variación en el número de setas abdominales en la mosca Drosophila pseudobscura frente al cambio de temperatura durante el desarrollo. La ausencia de paralelismo en las líneas muestra la interacción genotipo-ambiente (By Vbtbio - Own work, CC BY-SA 4.0, https://commons.wikimedia.org/w/index.php?curid=36851702).
La interacción entre genotipo y ambiente no solo se manifiesta en características cuantitativas. Por ejemplo, en el caso de patologías, la exposición a determinados elementos del ambiente puede hacer que genotipos que en otros ambientes no desarrollaban una enfermedad, en uno nuevo ambiente sí lo hagan, mientras que otros genotipos son insensibles a esto. En los seres humanos, los efectos ambientales son complejos y su importancia relativa puede variar considerablemente en el tiempo y depender de la genética de los individuos. Por otro lado, en muchos casos la interacción entre los factores físicos, los sociales y los económicos contribuyen en forma importante a la interacción GxE, muchas veces de formas complejas (como en el caso del tabaquismo, el alcoholismo y la malnutrición). Como resultado, la interacción GxE puede darse en la aparición o no de la enfermedad, en el tiempo para que aparezca, en el ritmo de su progresión y en la respuesta clínica a la intervención farmacológica.
Algunos ejemplos de estas interacciones entre genotipo y ambiente son, donde la combinación aumenta el riesgo de padecer enfermedades son las siguientes:
Pesticidas organofosforados y enfermedad de Parkinson
Si bien la enfermedad de Parkinson (EP) es del tipo multifactorial, con una combinación de influencias que incluyen variantes genéticas, exposición ambiental y estilo de vida, existen casos documentados de aumento sustantivo en el riesgo de desarrollar la enfermedad de Parkinson luego de la exposición organofosforados para genotipos específicos. En particular, el gen PON1 codifica una enzima paraoxonasa que metaboliza los organofosforados. Algunos individuos son portadores de una variante menos eficiente de PON1 y cuando se exponen a los organofosforados, tienen el doble de riesgo de contraer la enfermedad.
Cáncer de esófago y alcohol
El gen de la aldehído deshidrogenasa 2 (ALDH2) produce una enzima importante para el metabolismo del alcohol, con dos variantes diferentes en el gen, una que produce una enzima que funciona y otra (una mutación estructural generada por el intercambio del animoácido lisina por glutamato en la posición 487) que produce una enzima que no funciona. La mayoría de los caucásicos tienen dos isozimas principales, mientras que aproximadamente el 50% de los asiáticos orientales tienen una copia normal del gen ALDH2 y una copia de la forma inactiva. Las personas portadoras de la variante no funcional del gen ALDH2 tienen problemas importantes para metabolizar el alcohol, ya que la enzima defectuosa provoca la acumulación de aldehídos en el organismo (que se conoce como síndrome de enrojecimiento o “flushing”). Los individuos portadores (heterocigotos) tienen una reducción de alrededor de 100 veces en el metabolismo del alcohol. Estos heterocigotos tienen un mayor riesgo de padecer cáncer de esófago cuando se exponen a un consumo moderado o intenso de alcohol etílico en comparación con alguien que tiene ambas variantes funcionales.
Fenilcetonuria Se trata de una alteración congénita del metabolismo causada reducción o ausencia de la actividad en la enzima fenilalanina hidroxilasa, codificada por el gen PAH, localizado en el cromosoma 12. La misma tiene como cometido metabolizar el aminoácido fenilalanina (“Phe”). Se conocen más de 400 mutaciones en ese gen que producen una alteración en la capacidad metabólica del mismo, lo que lleva bajo una dieta normal a un incremento patológico en los niveles de fenilalanina. Sin embargo, en una dieta estrictamente controlada, con bajos niveles de fenilalanina la enfermedad no se manifiesta (en ninguno de los dos genotipos), lo que indica una interacción genotipo-ambiente.
En plantas, los programas de mejora de cultivos disponen cada vez más de datos genómicos, así como de rendimiento en el campo y medioambientales. El análisis de esta información de forma integrada puede facilitar la predicción genómica dentro y entre ambientes, así como proporcionar información sobre la arquitectura genética de rasgos complejos y la naturaleza de las interacciones genotipo-ambiente. Para entender la relaciones entre varianzas aditiva y de dominancia, así como los efectos de la interacción genotipo-ambiente, Rogers et al. (2021) recopilaron y analizaron los datos genotípicos y fenotípicos de 1918 híbridos de maíz (Zea mays L.) y los datos ambientales de 65 ambientes de prueba. Para el rendimiento del grano, la varianza de dominancia fue similar en magnitud a la varianza aditiva, y las varianzas GxE fueron más importantes que las varianzas genéticas de efecto principal.
Para otros rasgos, como el tiempo de floración, la varianza GxE sólo representó una pequeña parte de la varianza fenotípica, lo que sugiere que puede ignorarse con seguridad al realizar selección para estas caarcterísticas. Los resultados de este estudio sugieren que, para la predicción genómica de rendimiento de los híbridos en función del ambiente, la dominancia y las interacciones GxE deberían incorporarse a los modelos de predicción. Por otra parte, los modelos que incluían tanto los efectos aditivos como los de dominancia fueron los que mejor se ajustaron a los datos, y la modelización de las covarianzas genéticas únicas entre todos los ambientes proporcionó la mejor caracterización de los patrones de interacción genotipo-ambiente. A su vez, la similitud del rendimiento relativo de los híbridos entre los ambientes se modificó en función de las variables climáticas subyacentes, lo que permitió identificar las covariables climáticas que impulsan las correlaciones de los efectos genéticos entre los entornos.
PARA RECORDAR
- Debemos tener en cuenta que la interacción entre genotipo y ambiente no solo se manifiesta en características cuantitativas. Por ejemplo, en el caso de patologías, la exposición a determina- dos elementos del ambiente puede hacer que genotipos que en otros ambientes no desarrollaban una enfermedad, en uno nuevo ambiente sí lo hagan, mientras que otros genotipos son insensibles a esto.
15.6 Conclusión
Para finalizar esta sección (y el libro) nos adentramos en un tema de suma complejidad: la interacción genotipo-ambiente. Hasta este momento hemos considerado los componentes de la varianza como genéticos (aditivos) o ambientales, incluyendo en este último componente errores de medición y todo aquello que escapa a la herencia como la conocemos clásicamente. No obstante, como desarrollamos en este capítulo, la biología es compleja y un mismo genotipo puede mostrar diferentes fenotipos en ambientes disímiles. Esta interacción es difícil de modelar, y a su vez posee una importancia conceptual muy grande en campos distintos. A nivel práctico, resulta claro que es atractivo a nivel industrial buscar genotipos que sean lo más estables posibles en diferentes ambientes. Pero incluso para la ciencia básica tener en cuenta estos factores es sumamente importante: ¿podemos trasladar nociones obtenidas en el laboratorio a diferentes condiciones? ¿Cómo afecta la noción de plasticidad fenotípica a la evolución de las especies, siendo que este concepto escapa al componente hereditario clásico sobre el cual se suele parar la teoría evolutiva más sencilla? Adentrarse en estos problemas no es sencillo, ni a nivel matemático ni conceptual. Por lo tanto, este último capítulo puede ser entendido como una primera aproximación a un problema complejo. Lo que es más, podemos tomarlo de ejemplo para entender que, si nos adentramos lo suficiente en la complejidad de los temas planteados en los capítulos anteriores, la misma idea puede surgir. Con los conocimientos adquiridos en este libro, la persona interesada puede referirse a la bibliografía citada para comenzar a profundizar en la riqueza de dichos temas.
15.7 Actividades
15.7.1 Control de lectura
¿Qué es la plasticidad fenotípica? ¿Y la norma de reacción?
¿A qué refiere el concepto de canalización? ¿Cómo se vincula con el proceso evolutivo?
Desarrolle alguna aproximación para el estudio de la interacción genotipo-ambiente.
¿Qué estimador conoce del coeficiente de correlación genética (aditiva) entre ambientes (\(\rho_x\))?
Plantee ejemplos donde la interacción genotipo-ambiente posee un rol preponderante en el fenómeno biológico.
15.7.2 ¿Veradero o falso?
La presencia de interacción \(GxE\) necesariamente implica un re ranking de los genotipos.
La interacción entre genotipo y ambiente se manifiesta tanto en características cuantitativas como cualitiativas.
En caso de que la interacción genotipo-ambiente sea nula, la correlación genética entre ambientes es igual a cero.
Cuando el coeficiente de correlación genético (aditiva) estimado para dos características es \(\rho_x = 1\) no existe interacción genotipo-ambiente.
Si bien en cada ambiente un genotipo puede expresar un determinado fenotipo de manera óptima, existen rangos ambientales donde las respuestas poblacionales son aproximadamente equivalentes en diferentes ambientes.
Soluciones
Falso. Puede existir un cambio de escala sin re-ranking.
Verdadero. Podemos pensar en las enfermedades como un caso claro donde la interacción genotipo-ambiente puede llevar a efectos cualitativos: la presencia de ciertos metabolitos puede inducir cambios en estados celulares que lleven a una patología, y la respuesta dada por un genotipo puede variar para un rango dado del factor ambiental, manifestándose la patología solo luego de cierto umbral. El mismo razonamiento lleva a pensar también en casos como el desarrollo de determinadas morfologías, donde el pasaje de un determinado punto de corte puede inducir una respuesta celular que a través de una cascada lleve al desarrollo de un caracter.
Falso. Si el coeficiente es igual a uno se trata de la misma característica (pese a haberla considerado como dos características diferentes para el análisis).
Verdadero. En efecto, si \(\rho_x = 1\) lo que estamos considerando como dos características son en efecto una. Por lo tanto, no puede existir interacción genotipo-ambiente en este caso
Veradero. Tomemos como ejemplo la respuesta a la disponibilidad hídrica en especies de plantas para ilustrar este concepto. Es posible notar que más allá que cada genotipo tenga un óptimo de producción, generalmente los extremos (anegamientos o sequías prolongadas) generarán que ambos genotipos tengan el mismo comportamiento. Si bien desde el punto de vista cuantitativo los óptimos pueden variar levemente, desde una visión más biológica es razonable pensar que las respuestas serán razonablemente parecidas en los rangos ambientales medios entre los extremos planteados.
15.7.3 Ejercicios
 Ejercicio 15.1
Ejercicio 15.1
A continuación se presenta el comportamiento en rendimiento (kg/ha) de dos híbridos de maíz bajo diferente disponibilidad hídrica en el rango de \(30\) a \(80%\) de agua disponible (AD).
a. Desarrolle un modelo lineal que describa el comportamiento de cada híbrido frente a la variación de disponibilidad hídrica. b. ¿Cuál de los híbridos tiene mayor respuesta al aumento en la disponibilidad hídrica?
Solución
a. Valiéndonos del uso de una herramienta de cálculo (planilla Excel, etc.) podremos graficar y posteriormente obtener el modelo lineal asociado a cada híbrido como se observa a continuación.
Como se observa, el modelo lineal que mejor predice el comportamiento del híbrido 1 es Rend(kg/ha)\(=1439 + 71,714 %AD\) mientras que para el híbrido \(2\) es Rend(kg/ha)\(= -1744,2 + 124,74 %AD\).
b. El híbrido con mayor respuesta al aumento en la disponibilidad de agua es el \(2\) debido a que su rendimiento aumenta casi \(125\) kg/ha por cada unidad de aumento en el %AD.
 Ejercicio 15.2
Ejercicio 15.2
Dos genotipos de avena negra (Avena strigosa) presentan diferente respuesta a la temperatura en el rango entre \(7\) y \(30\text{ ºC}\), lo que afecta su capacidad de generar biomasa foliar. La variedad A posee un rendimiento en \(\text{kg/ha}\) que puede ser descrito por la función \(A= 3200 − (t − 22)^2\text{ kg/ha}\) mientras que para la variedad B sería \(B=2900+15t\).
¿A qué valor o valores de temperatura ambas variedades producen la misma biomasa foliar?
Solución
Para evaluar a qué temperaturas ambas variedades producen lo mismo deberemos igualar las funciones que describen el comportamiento de cada una:
\[[3200-(t-22)^2]=[2900+15\ t\ ] \Leftrightarrow 3200-2900=(t-22)^2+15t \Leftrightarrow 300=t^2-44t+22^2+15t \Leftrightarrow t^2-29t+184=0\]
Obtuvimos como resultado una ecuación cuadrática, y revolviéndola obtendremos las dos raíces que representan las intersecciones entre ambas funciones:
\[ \begin{split} t^2-29t+184=0 \Leftrightarrow t=\frac{29 \pm \sqrt{29^2-4\cdot 184}}{2}\Leftrightarrow \\ t=\frac{29\pm \sqrt{105}}{2} \Leftrightarrow t=\frac{29 \pm 10,2}{2} \Leftrightarrow \\ t=9,4^{\circ}\text{C};\ t=19,6^{\circ}\text{C} \end{split} \]
Por lo tanto, ambas variedades producirán la misma cantidad de biomasa a los \(9,4\) y a los \(19,6\ ºC\).
 Ejercicio 15.3
Ejercicio 15.3
Un grupo de investigación se dispone a estudiar la interacción genotipo-ambiente de una especie que crece en un rango de temperaturas amplio. En las poblaciones estudiadas encuentran tres genotipos para un locus dado, el cual se ve correlacionado con la capacidad metabólica a diferentes temperaturas. El alelo \(A\) es dominante, por lo que el grupo decide contrastar el comportamiento de organismos de genotipo \(A./A.\) respecto al de organismos homocigotas recesivos \(aa/aa\).
Luego de un arduo trabajo, determinan que la capacidad metabólica (medida por satélite como producción de \(CO_2\) por área del organismo, \(g \cdot cm^{-2} CO_2/m^3\)) sigue las siguientes ecuaciones de dependencia con la temperatura (T, en grados Celsius)
\[ \begin{cases} \text{Capacidad metabólica (A./A.) (T)} = [−T^2 +61,2T−812,55] g \cdot cm^{-2} CO_2/m^3 \\ text{Capacidad metabólica (aa/aa)} = [1.5T + 2] g \cdot cm^{-2} CO_2/m^3 \end{cases} \]
¿Reaccionan igual ambos genotipos frente al ambiente?
Solución
Claramente ambos genotipos no reaccionan de forma similar al ambiente: una de las normas de reacción sigue una ecuación de segundo grado (parábola), mientras que la otra una ecuación lineal. Esto constituiría un caso bastante peculiar de interacción genotipo-ambiente.
Si graficamos ambas normas de reacción obtenemos el siguiente gráfico
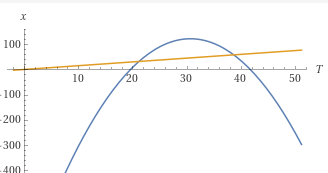
Observamos que existe un rango inicial en el cual el Genotipo 2 posee mayor capacidad metabólica que el Genotipo 1. La situación se revierte, pero a medida que aumenta la temperatura la capacidad metabólica de este último genotipo alcanza un pico, volviendo hacia el final a ser superada por la capacidad del Genotipo 1.
 Ejercicio 15.4
Ejercicio 15.4
En un laboratorio se estudia la norma de reacción frente a la temperatura de dos fenotipos (largo de ala y largo de antenas) en individuos de D. melanogaster. El grupo de investigación sabe de la literatura que dos genotipos son la base de cada genotipo observado en cada caso, por lo que se disponen a analizar dos grupos de individuos de líneas puras para rangos de temperaturas controladas en cada caso.
Un análisis estadístico arroja los siguientes datos:
Largo de ala
\[ \begin{cases} \sigma^2_T = 0,39 \\ \sigma^2_{largo} = 0,67 \\ \sigma_{T,largo} = 0,09 \end{cases} \]
Largo de antenas
\[ \begin{cases} \sigma^2_T = 0,34 \\ \sigma^2_{largo} = 0,45 \\ \sigma_{T,largo} = 0,149 \end{cases} \]
¿Existe interacción genotipo-ambiente en alguno de estos casos?
Solución
Recordemos que podemos estimar la correlación de las medidas de temperatura y cada fenotipo para los grupos genotípicos según
\[ \begin{split} \rho_x=\frac{\sigma_G(1,2)}{\sigma_G^2(1)\ \sigma_G^2(2)} \end{split} \]
Tenemos así que para el caso del largo de alas
\[ \begin{split} \rho_x=\frac{0,09}{0,39 \cdot 0,67} \approx 0,34 \end{split} \]
esta correlación se encuentra claramente alejada de un valor absoluto de 1, lo cual sugiere la existencia de interacción genotipo-ambiente en este caso.
De forma análoga, tenemos para el caso del largo de antena
\[ \begin{split} \rho_x=\frac{0,149}{0,34 \cdot 0,45} \approx 0,97 \end{split} \]
En el caso de largo de antena observamos que la estimación de correlación obtenida es altísima, a efectos prácticos considerable en 1. Esto no permite sugerir la existencia de una interacción genotipo-ambiente en el caso de este caracter.
 Ejercicio 15.5
Ejercicio 15.5
(Inspirado en Feldman and Lewontin 1975)
Supongamos que varios loci influencian a un caracter fenotípico que controla la producción de leche de un mamífero en poblaciones naturales, teniendo una altísima heredabilidad. La norma de reacción de los dos genotipos existentes frente a la temperatura (en grados Celsius) es la siguiente
\[ \begin{cases} \text{Genotipo 1}: y = [0,5T + 12] L/día \\ \text{Genotipo 2}: y = [-0,5T + 36,2] L/día \\ \end{cases} \]
Para sorpresa de un grupo de investigadores, un estudio del caracter fenotípico a lo largo de un área estudiada muestra un promedio estable, a pesar de que se observa variación en el caracter fenotípico. ¿Cómo es esto posible, y cual es el valor medido por los investigadores?
Pista: Es conveniente graficar ambas normas de reacción para observar su comportamiento.
Solución
A continuación se observa el resultado de graficar ambas normas de reacción
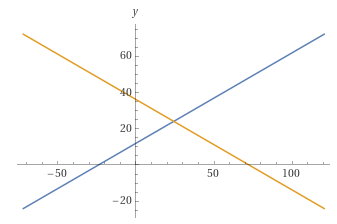
Como se puede observar, ambas normas de reacción parecen simétricas respecto a un punto de intersección. ¿Qué implica esto? A medida que cambia un rango de temperaturas, uno de los genotipos aumentará el valor del caracter fenotípico exactamente lo mismo que lo disminuye el otro. Si en una población se observa el reemplazo de un genotipo por el otro, pero esto se ve acompañado por una disminución del caracter fenotipico (producción de leche), entonces el promedio en la población se mantendrá estable aún cuando varíe el genotipo.
El valor en el que se debería encontrar este promedio que se mantiene estable se obtiene de igualar ambas ecuaciones. Tenemos
\[0,5T + 12 = - 0,5T + 36,2\] \[1T = 36,2 - 12\] \[T = 24,2\]
por lo que estimamos que los investigadores miden aproximadamente \(24,2\) litros de leche por día en cada animal lactante.