Capítulo 1 Introducción a la Genómica
Ejercicio 1.1
Luego de un muestreo realizado a partir de metagenómica en dos ambientes diferentes, la distribución de especies (OTUs) en los mismos es la siguiente
| Especie | Ambiente 1 | Ambiente 2 |
|---|---|---|
| OTU 1 | \(400\) | \(1\) |
| OTU 2 | \(150\) | \(700\) |
| OTU 3 | \(0\) | \(297\) |
| OTU 4 | \(390\) | \(1\) |
| OTU 5 | \(60\) | \(1\) |
| Total | \(1000\) | \(1000\) |
Calcular los diferentes índices de diversidad alfa para los dos ambientes (Shannon, Simpson y Good) y discutir los resultados.
Ejercicio 1.2
Con la internacionalización del comercio, y en especial con la globalización, venimos observando una dramática degradación de la biodiversidad que ha llevado a la pérdida de decenas de razas adaptadas a las condiciones locales. El principal problema que enfrenta la conservación de dichas razas es mantener un número suficientemente elevado para reducir las pérdidas por endogamia, al tiempo de evitar la introgresión de material genético por parte de otras razas, todo esto en un contexto de viabilidad económica. En un esfuerzo por mantener la diversidad en bovinos, un gobierno lanzó un programa de subvenciones para las regiones que mostrasen un esfuerzo sostenido en tal sentido. El límite de animales a subvencionar se estableció en 10 mil para cada región.
Tres regiones (subdivisión política aceptada por dicho gobierno) se presentaron al programa, cada una con diferentes números para diferentes razas, pero todas presentaron el máximo de 10 mil animales, como lo muestra la tabla siguiente:
| Raza | Región 1 | Región 2 | Región 3 |
|---|---|---|---|
| Randall Lineback Cattle | \(1400\) | \(520\) | \(2800\) |
| Dutch Belted | \(2250\) | \(700\) | \(3500\) |
| Red Devon | \(0\) | \(2970\) | \(0\) |
| Galloway | \(5800\) | \(3800\) | \(2600\) |
| Ankole-Watusi | \(0\) | \(140\) | \(0\) |
| Lincoln Red | \(490\) | \(900\) | \(0\) |
| Kerry | \(60\) | \(970\) | \(1100\) |
| Total | \(10000\) | \(10000\) | \(10000\) |
El responsable del programa de conservación debe decidir cuánto del “budget” anual destinado a subsidios (U$S 15 millones) se le debe destinar a cada región.
Teniendo en cuenta los índices de diversidad alfa Shannon y Pielou, realizar una propuesta (para cada índice) de pago razonable para los tres regiones y discutir los resultados. ¿Cuánto dinero por animal recibe cada región como subvención en cada una de las propuestas? ¿Resulta un mecanismo atractivo para la conservación de la diversidad in-situ?
¿Qué diferencias y problemas se encontrarían asociados al pago por el índice de Pielou?
¿Cuál podría ser un problema importante de considerar todas las razas en pie de igualdad (todas pesan de la misma manera en el programa) y cuál podría ser una solución a este problema?
Ejercicio 1.3
En la gráfica siguiente se observa el comportamiento del índice de Shannon-Wiener a medida de que aumenta el número de especies (razas) en la muestra, todas ellas con el mismo número de individuos (por ejemplo, una muestra de 1000 individuos, con 10 especies, cada una de ellas con 100 individuos).
Describir en términos cualitativos el comportamiento del índice en este caso.
Dado de que en este caso la uniformidad es máxima, ¿qué representa el índice de Shannon-Wiener en este caso particular?
Demostrar que en este caso particular, el valor del índice es \(H'=ln(k)\), con \(k\) el número de especies.
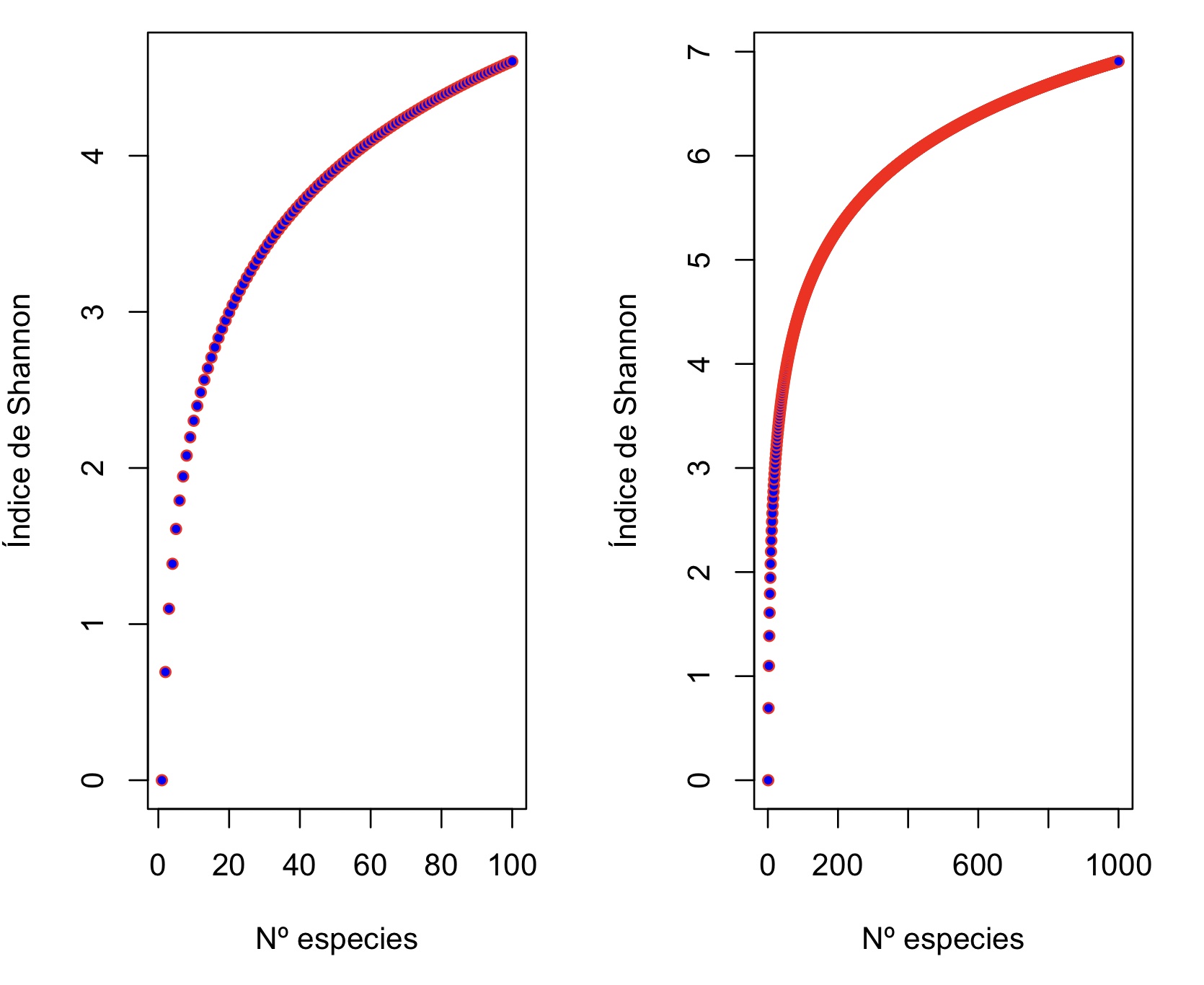
Figure 1.1: Valor del Índice de Shannon-Wiener a medida de que aumenta el número de especies (o razas), todas con el mismo número de individuos.
Ejercicio 1.4
A partir del ensamblado primario de una secuenciación de los transcriptos de una bacteria (RNAseq) se observó el siguiente conteo de codones para los aminoácidos Serina y Alanina:
| Aminoácido | Codón | Conteo |
|---|---|---|
| Ser (S) | UCU | \(666\) |
| Ser (S) | UCC | \(1320\) |
| Ser (S) | UCA | \(1457\) |
| Ser (S) | UCG | \(1818\) |
| Ser (S) | AGU | \(5913\) |
| Ser (S) | AGC | \(5120\) |
| Ala (A) | GCU | \(7123\) |
| Ala (A) | GCC | \(2144\) |
| Ala (A) | GCA | \(1125\) |
| Ala (A) | GCG | \(8213\) |
Calcular el RSCU para cada codón y determinar cuáles son los más usados y si hay diferencias entre los aminoácidos en la forma en que usan los codones.
Ejercicio 1.5
Dada la siguiente secuencia “AUGGGAUUUCCGCCCAUUUACGUG”,
- determinar de que ácido nucléico se trata (ADN o ARN),
- si tiene sentido, traducirla a una secuencia de aminoácidos tomandola como hebra codificante y en marco de referencia, y
- escribir en sentido 5’3’ la hebra complementaria pero en ADN (hebra molde) d) calcular el contenido G+C.
Ejercicio 1.6
A fin de diseñar un chip de genotipado para estudiar la variabilidad genética en Paspalum dilatatum se extrajeron 8 secuencias de ADN disponibles en la base de datos “Nucleotide” de GenBank (https://www.ncbi.nlm.nih.gov/nuccore/?term=Paspalum+dilatatum). Luego de alinear estas secuencias se detectaron en total 12 sitios (S) (algunos de los cuales son polimórficos en un sólo nucleótido, SNP) que se indican en la siguiente tabla:
| Secuencia | S1 | S2 | S3 | S4 | S5 | S6 | S7 | S8 | S9 | S10 | S11 | S12 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Secuencia 1 | A | C | C | G | A | T | C | T | C | G | A | A |
| Secuencia 2 | A | C | C | A | A | T | C | A | G | G | C | C |
| Secuencia 3 | C | C | C | C | A | T | C | T | G | G | C | A |
| Secuencia 4 | A | T | C | G | A | T | C | T | C | G | C | A |
| Secuencia 5 | A | C | C | G | A | T | C | T | C | G | C | A |
| Secuencia 6 | A | T | C | A | G | T | C | T | C | G | C | A |
| Secuencia 7 | A | C | C | T | G | T | C | T | C | G | C | C |
| Secuencia 8 | A | C | C | G | A | T | C | A | C | G | A | A |
Teniendo en cuenta esta tabla,
- ¿Qué sitios son polimórficos (despreciando el criterio de la frecuencia mínima)?
- De estos sitios ¿Cuáles son bi-alélicos?
- Utilizando el criterio de diversidad de Shannon (el mismo índice de Shannon que se utiliza en ecología, pero que originalmente viene de la teoría matemática de comunicaciones), calcular qué sitio es más informativo (más “diverso”). ¿Existe alguna limitación en la práctica para incluirlo en los chips?
Ejercicio 1.7
La siguiente es la secuencia de nucleótidos correspondiente al CDS completo del gen “cinnamoyl CoA reductase” (CCR1-3) de Paspalum dilatum, una enzima que desempeña un papel fundamental en la formación de lignina, un proceso importante en las plantas tanto para el desarrollo estructural como para la respuesta de defensa:
ATGACCGTCGTCGACGCCGTCTCGTCCGCCGCCGCCGTGGCGCAGCCGGCGGGGAACGGGCAGAC CGTGTGCGTCACCGGCGCGGCGGGGTACATCGCGTCGTGGCTCGTCAAGCTGCTGCTCGAGAAGG GGTACACTGTGAAGGGCACCGTGAGGAACCCAGATGACCCGAAGAACGCGCACCTCAGGGCGCTG GAGGGCGCCGCCGAGCGGCTGATCCTCTGCAAGGCCGACCTGCTGGACTACGACGCCATCTGCCG CGCCGTGCAGGGCTGCCAGGGCGTCTTCCACACCGCCTCCCCCGTCACCGACGATCCCGAGCAAA TGGTGGAGCCGGCGGTGCGCGGCACGGAGTACGTGATCAGCGCGGCGGCGGAGGCCGGCACGGTG CGGCGGGTGGTGTTCACGTCCTCCATCGGCGCCGTCACCATGGACCCCAACCGCGGCCCCGACGT CGTCGTCGACGAGTCCTGCTGGAGCGACCTCGAGTTCTGCAAGAAAACCAGGAACTGGTACTGCT ACGGCAAGGCGGTGGCGGAGCAGGCGGCGTGGGACGCGGCGCGGCACCGGGGCGTGGACCTGGTG GTGGTGAACCCGGTGCTGGTGGTGGGGCCGCTGCTGCAGCCGACGGTGAACGCCAGCATCGGGCA CGTCCTCAAGTACCTGGACGGCTCCGCGCGCACCTTCGCCAACGCCGTGCAGGCGTACGTCGACG TCCGCGACGTCGCCGACGCGCACGTCCGCGTCTTCGAGAGCCCCCGCGCCGCCGGCCGCCACCTC TGCGCCGAGCGCGTGCTCCACCGCGAGGACGTCGTCGACCTCGCCAAGCTCTTCCCCGAGTACCC CGTCCCGACCAGGTGCTCCGACGAGGTGAACCCGCGGAAGCAGCCGTACAAGTTCTCGAACCAGA AGCTCCGGGACCTGGGGCTGGAGTTTCGGTCGGTGAGCCAGTCGCTGTACGACACGGTGAAGTGC CTCCAGGAGAAGGGCCACCTGCCGGTGCCGGTGCCCGACGAGCAGACGGCGACGGAGGCCAAGAA GAAGACAGTGGCCCCCGCCGCCGAGCTGCAGCAGGGAGGAATCGCCATCCGTGCGTGA
- Traducir los 9 primeros codones de dicha secuencia.
- ¿Cuáles son los codones de inicio y terminación?
- Utilizando la función Reemplazar de OpenOffice, Word o cualquier programa similar contar cada una de las bases y calcular el contenido GC del gen.
- ¿Qué largo tiene el mRNA? ¿Para cuántos aminoácidos codifica?
- A partir de la siguiente tabla de uso de codones, determinar qué aminoácido de los que estos codifican es el más frecuente en este gen.
| aca | acc | acg | act | cca | ccc | ccg | cct | gca | gcc | gcg | gct |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 10 | 7 | 1 | 1 | 9 | 11 | 0 | 0 | 25 | 22 | 0 |
Ejercicio 1.8
Un investigador obtuvo la siguiente secuencia (según él) de un nuevo gen que sería responsable de la generación de radiactividad por parte de una especie bacteriana que lo posee (presentamos la secuencia ordenada en tripletes y en marco de lectura correcto, para mayor facilidad de visualización):
GTC TCT CGT GAT GCG GTT GTC ATA AGA GCT GGC TCT GAG CTG AAT TGA AGC ATA TCC TTC GCA GTC TTA AAA GGG TTG GGA TTA TCA ACC TCC CAC GCA ATA CCG GCG TTC AAC AGT TTA CCA TCA TCT ACT GAC GCG TGA TGT TGC GGA GCG ACG GGA AGT GAT AAA AGT TAC TTT ACA
La especie bacteriana de la que supuestamente fue aislado el gen, posee un contenido GC del \(80\%\).
Por otra parte, un estudiante de su grupo afirma que no solamente no se trata de la secuencia de un nuevo gen, sino que no se trata de la secuencia de ningún gen. Más aún, plantea que esta secuencia fue generada en forma aleatoria por una máquina y que posiblemente el investigador ha sido engañado por alguien. El conteo de bases dio lo siguiente:
| A | C | G | T |
|---|---|---|---|
| 46 | 40 | 45 | 49 |
mientras que el conteo de aminoácidos fue el siguiente:
| * | A | C | D | E | F | G | H | I | K | L | N | P | R | S | T | V | Y |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 2 | 7 | 2 | 3 | 1 | 3 | 5 | 1 | 3 | 2 | 5 | 2 | 2 | 2 | 11 | 4 | 4 | 1 |
¿Quién de los dos parece tener razón? Argumenta las razones que te llevan a esa conclusión.
¿Cuántas Cisteínas hay codificadas en la secuencia y dónde se encuentran?
¿Cuántas Serinas y cuántas Argininas serían de esperar en una secuencia de este largo si se tratase de una secuencia creada al azar y cuántas fueron observadas? ¿Y la suma de las dos (es decir, para los codones de los sextetos)?
Determinar mediante un test estadístico si el número de Serinas y de Argininas observadas cae dentro de lo esperado, o si debemos rechazar dicha hipótesis nula.
Ejercicio 1.9
Señala cuáles de las siguiente afirmaciones son verdaderas y cuáles falsas:
La metagenómica es el estudio mediante técnicas genómicas de los organismos productores de metano.
La transcriptómica es el conjunto de técnicas y métodos para el estudio de los niveles de abundancia de transcriptos (RNAm) en diferentes muestras.
El ARNr 16S es usado frecuentemente para estudiar la diversidad en eucariotas debido a que se trata de una molécula relativamente conservada.
El contenido GC (o G+C) de un organismo en la proporción de los aminoácidos Glicina (G) y Cisteína (C) en las secuencias codificantes del mismo.
Las proteínas integrales de membrana (IMPs) se distinguen de otras proteínas debido a que presentan un exceso de residuos hidrofílicos, lo que les permite ubicarse comodamente en la bicapa lipídica.
Las proteínas solubles en agua suelen poseer los residuos hidrofóbicos (Leu, Ile, Val, Phe y Trp) escondidos en el medio de la proteína (en su estructura), mientras que los hidrofílicos se encuentran expuestos en la superficie.
Usualmente las secuencias ortólogas son las más similares en las otras especies y esto ha llevado al criterio práctico del “Reciprocal Best Hit”, es decir, un par de secuencias se consideran ortólogas si cada una de ellas es la mejor “selección” (la más similar) en su correspondiente genoma, de la otra.
Algunas variables biológicas asociadas a la variación en el uso de codones dentro de genomas procariotas son el contenido GC del gen, el nivel de expresión, la hidropatía promedio de la proteína, la hebra en que se encuentra el gen, el GCskew y la precisión en la traducción.
La epigenética se centra en los procesos que regulan la activación y desactivación de genes, tanto en lo que hace a cómo y cuándo se produce esto.
Las tecnologías de secuenciación masiva (Illumina, ION Torrent, Oxford Nanopore Technologies, PacBio) son las que se emplean en los chips de genotipado que se usan comercialmente.
En los chips de genotipado se suelen utilizar marcadores bi-alélicos porque son los más informativos.
En el código genético hay 64 codones.
El código genético universal hay 22 aminoácidos codificados directamente, además de 3 codones destinados a la señal de pare (codones STOP).
Los 64 codones del código genético universal se corresponden a los 3 codones destinados a la señal de pare (codones STOP), además de para los 61 aminoácidos proteinogénicos.
Ejercicio 1.10
Demostrar que para cualquier codón de cualquier aminoácido, el RSCU esperado es de 1.
Ejercicio 1.11
Observando la tabla con el código genético universal agrupar los aminoácidos por el número de codones que los codifican y resumir el número de aminoácidos con cada multiplicidad. ¿Qué característica comparten la mayor parte de los duetos (los aminoácidos codificados por dos codones)?
Ejercicio 1.12
En un planeta de otra galaxia existe vida con algunas similitudes a la que existe en nuestro planeta. En particular, también las proteínas parecen ser la base estructural y funcional de la vida, mientras que los ácidos nucléicos son los responsables del mantenimiento y duplicación de la información. Sin embargo, a diferencia de nuestro planeta, los aminoácidos proteinogénicos codificados son 24, mientras que las bases nucleótidicas análogas a las de nuestro ADN son 5.
Suponiendo que los mecanismos moleculares en dicho planeta han sido seleccionados para ser altamente eficientes,
- ¿de qué largo mínimo deberían ser los codones?
- Si el código genético en ese planeta tuviese algún nivel de redundancia (más de un codón para el mismo aminoácido), ¿de qué largo mínimo serían los codones? En ese caso, ¿cuál sería el número promedio de codones por aminoácido?
- ¿Cómo se compara esa redundancia promedio con la existente en nuestro planeta?
- ¿Qué ventaja potencial tiene la redundancia en el código?
Ejercicio 1.13
Asumiendo que existen 20 aminoácidos (los estándar),
- ¿Cuántas proteínas diferentes de largo 5 aminoácidos sería posible tener?
- ¿Cuántas serían posibles de largo \(n\)?
- Considerando que una proteína de largo promedio en procariotas es del orden de los 300 aminoácidos, ¿cuántas proteínas diferentes, con ese largo se podrían codificar?